simplifyEnrichment,一个对GO富集结果进行聚类和可视化的工具
在前一篇博文中,我介绍了我的一个新的Bioconductor包cola,在这篇博文中,我继续介绍另一个新的R包simplifyEnrichment。Bioconductor上的链接为https://bioconductor.org/packages/simplifyEnrichment/,论文链接为https://www.biorxiv.org/content/10.1101/2020.10.27.312116v1。
对基因的功能富集分析是生物信息学中的一个基本的分析方法,通常用户会得到成百上千个显著富集的功能。那么下一步就是如何将这成百上千个功能减少到一个适当的值,这样用户可以很方便的阅读,并且不会丢失重要的信息。
simplifyEnrichment主要针对于GO富集分析的结果,为了对富集出来的GO列表进行简化,我们首先要得到GO和GO之间的相似性,然后基于此,把GO划分到几个类中。一般来说,我们用基于语义学的GO相似性度量,例如使用GoSemSim包,在得到GO相似性矩阵后,simplifyEnrichment提供了一个新方法,称之为binary cut,用来对GO相似性矩阵进行划分。
下图是一个对GO相似性矩阵进行划分后的结果,同时对每个GO cluster,我使用word cloud作为对应的annotation,这样可以很容易的知道每个GO cluster所对应的功能。
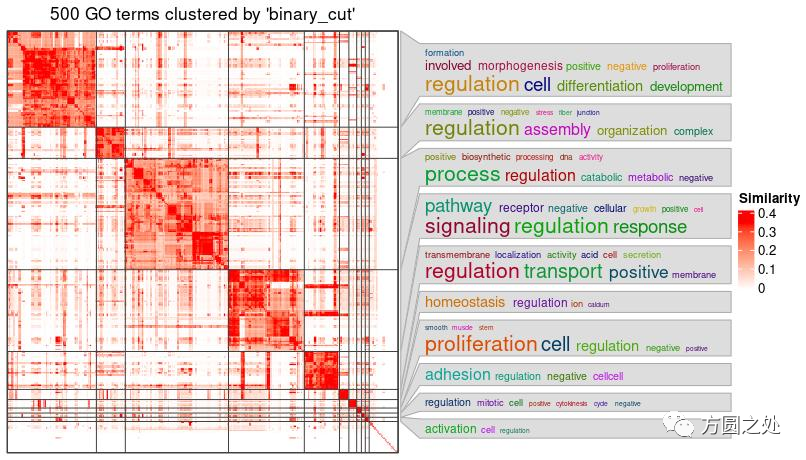
simplifyEnrichment的使用方法也很简单,用户提供一个GO列表,使用GO_similarity()函数计算相似性矩阵,然后使用simplifyGO()对GO进行聚类并生成图。
library(simplifyEnrichment)
mat = GO_similarity(go_id)
df = simplifyGO(mat)
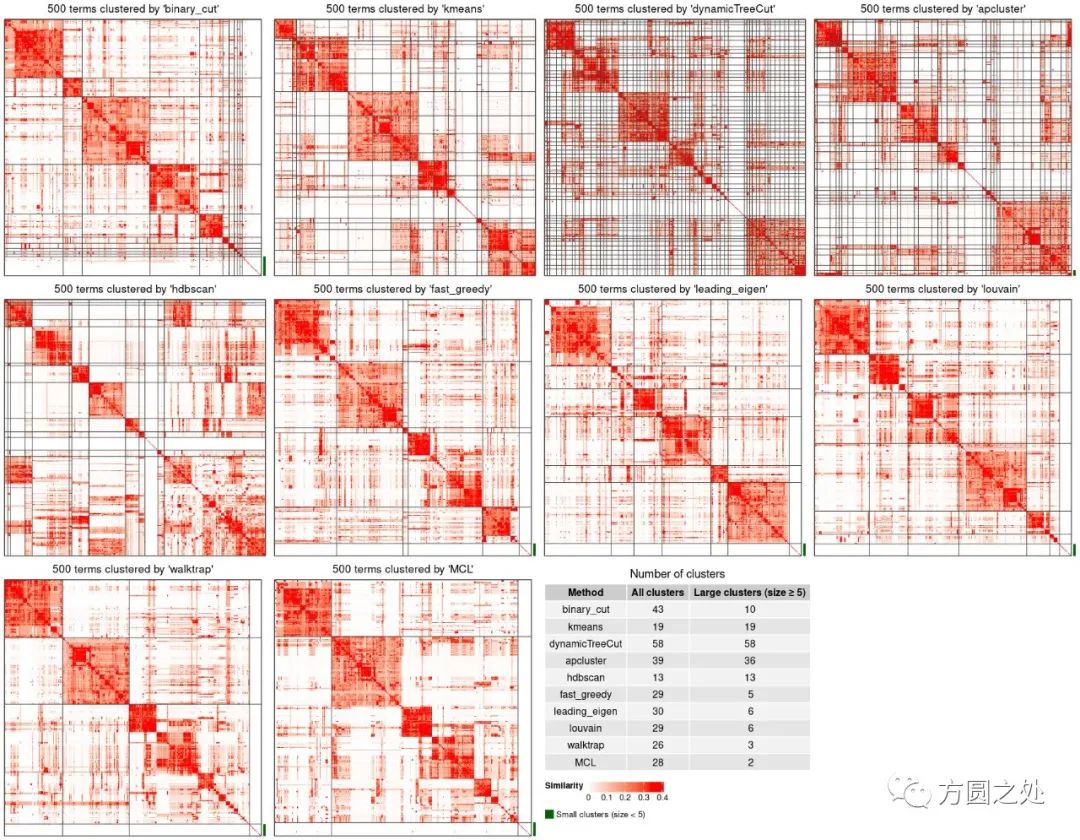
对GO相似性矩阵进行聚类看似是一个简单的问题,其实在实践中会存在几个问题,使得某些相似GO无法被聚在一起,或者一个较大的GO类中的异质性还是很高。下面一张图显示了一些不同的聚类方法对同一个GO相似性矩阵进行划分的结果,可见,一些方法生成了太多的聚类(如第一行第三列),而另外一些方法无法将大的聚类划分为更小的聚类(如第三行第二列)。相比而言,binary cut (第一行第一列)能够很好的将GO划分为类,并且同时能够划分出大的聚类和小的聚类。
这个只需一步就可做富集分析的网站还未发表就被CNS等引用超过350次
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
(请备注姓名-学校/企业-职务等)



























