中山大学骆观正教授 | 核酸修饰的鉴定和编辑技术前沿综述 (全文翻译...

核酸修饰是指在DNA或RNA碱基上引入额外的化学基团的修饰方式,它不改变基因序列的排列方式却极大地提高了碱基的编码遗传信息的能力。近年来,核酸修饰在基因表达调控中所起的作用越来越受到人们的关注。被修饰的碱基作为DNA或RNA上的标记物被特定的蛋白质所识别,参与了基因表达调控并进一步影响了生理功能。然而,碱基修饰通常不改变碱基的配对方式,所以难以被PCR等分子生物学手段直接鉴别。多年以来,方法学的欠缺严重阻碍了核酸修饰研究的发展。最近,随着新技术的不断进步,DNA和RNA上多种修饰的准确鉴定和定位问题有了突破。同时,在基因编辑工具的启发下,研究人员通过整合基因靶向工具和修饰相关的效应蛋白开发了一系列新型的核酸修饰编辑工具,尝试改变目的基因的修饰状态来研究特定位点的核酸修饰机制。
2020年3月19日,中山大学骆观正教授实验室在Computational and Structural Biotechnology Journal在线发表了文章“Mapping and editing of nucleic acid modifications”。在这篇综述中,总结了DNA和RNA修饰的特点以及现有的检测技术和编辑工具,以及这些技术和工具在基础和转化医学研究中的潜在应用和现有不足。
核酸修饰的鉴定和编辑技术前沿
作者 | 陈丽倩 赵文烁 骆观正*
中山大学生命科学学院,有害生物控制与资源利用国家重点实验室,基因功能与调控教育部重点实验室
通讯地址:luogzh5@mail.sysu.edu.cn
摘要
核酸修饰是指在DNA或RNA碱基上引入额外的化学基团的修饰方式,它不改变基因序列的排列方式却极大地提高了碱基的编码遗传信息的能力。近年来,核酸修饰在基因表达调控中所起的作用越来越受到人们的关注。被修饰的碱基作为DNA或RNA上的标记物被特定的蛋白质所识别,参与了基因表达调控并进一步影响了生理功能。然而,碱基修饰通常不改变碱基的配对方式,所以难以被PCR等分子生物学手段直接鉴别。多年以来,方法学的欠缺严重阻碍了核酸修饰研究的发展。最近,随着新技术的不断进步,DNA和RNA上多种修饰的准确鉴定和定位问题有了突破。同时,在基因编辑工具的启发下,研究人员通过整合基因靶向工具和修饰相关的效应蛋白开发了一系列新型的核酸修饰编辑工具,尝试改变目的基因的修饰状态来研究特定位点的核酸修饰机制。在这篇综述中,我们总结了DNA和RNA修饰的特点以及现有的检测技术和编辑工具,以及这些技术和工具在基础和转化医学研究中的潜在应用和现有不足。引言
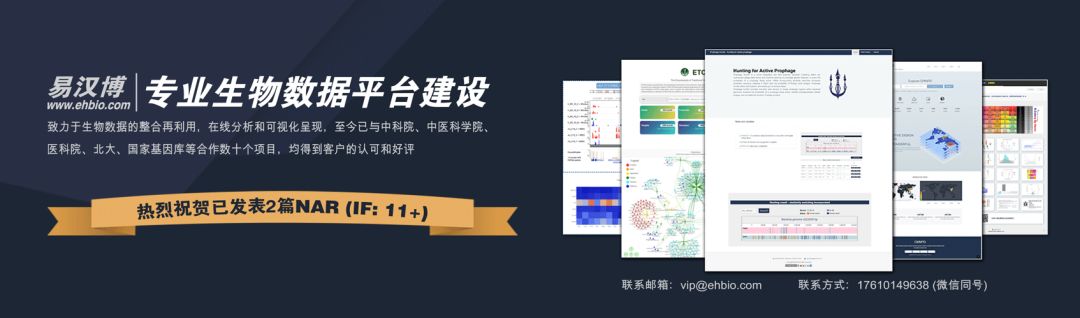
经典的中心法则中遗传信息通常是从DNA流向RNA,然后再以蛋白质的形式表达。然而,除了遗传密码之外,核酸还蕴藏了另外一层复杂的由化学修饰主导的表观遗传修饰系统来扩展自身信息编码的能力[1]。这些修饰通过调节基因表达网络参与了多种重要的生物学过程,也被叫做表观遗传修饰[2]。表观遗传修饰通常包括DNA、RNA和蛋白质上发生的化学修饰,而我们将DNA和RNA上的化学修饰称之为核酸修饰。核酸修饰调节基因表达不是通过改变遗传密码,而是通过“阅读”蛋白(‘reader’ proteins)和相关的效应蛋白(“编写器”和“擦除器”蛋白,‘writer’和 ‘eraser’ proteins)来起作用的[3]。到目前为止,已经先后发现了几十种不同的DNA修饰和上百种不同的RNA修饰(主要类型如图1所示) [4,5]。这些修饰在“编写器”蛋白或“擦除器”蛋白作用下在核酸上进行添加或者去除化学修饰基团,并被作为“阅读器”的蛋白进行识别发挥功能[6]。近期有越来越多的研究表明,多种DNA和RNA修饰主动参与了多种生命过程的调控,包括胚胎发育、器官分化等。这些修饰失调有可能会导致人类疾病,甚至死亡[7]。随着修饰研究的深入,越来越多修饰的相关效应蛋白被发现,在此基础上,研究者们试图将其与最新的基因编辑工具联合起来开发新的分子工具,希望通过在特定位点建立或去除修饰,更精确地研究这些修饰的生物学功能和相关调控机制[8]。
然而,核苷酸修饰的研究多年来一直进展缓慢,其中发展受限一个主要原因是很多修饰的丰度相对较低,使得大多数修饰很难检测和研究。例如,5mC作为哺乳动物中最丰富的DNA修饰,约占总DNA碱基的1%[9], 而mRNA上最丰富的修饰m6A,仅占所有腺苷碱基的0.1-0.4% [10],其他类型的修饰比例则更低[5]。此外,这些微量的碱基修饰是高度动态的,有时是可逆转的,会随着生理条件的改变或发育阶段而异[11]。因此,积极开发创新方法来检测和定位它们的精确位点是研究这些难以捉摸的修饰的关键[12]。然而,每种类型的修饰都有其独特的属性,这极大地限制了通用方法的开发和运用。伴随着高通量测序等先进技术的快速发展,各种修饰的定位也都逐渐被破解 [13]。与此同时,研究人员也开始寻找可以改变目的基因的修饰状态的新工具,尽管目前每种修饰的效应蛋白各有不同,并且缺乏详细的特性描述,但是这些新工具的开发也在很多生物问题上为修饰领域提供了新的理解角度[14,15]。为了更好理解核酸修饰的多样性和复杂性,我们总结了目前关于核酸代表性修饰类型的知识和相关技术,并强调了精准定位和编辑工具在这一研究领域的重要性。
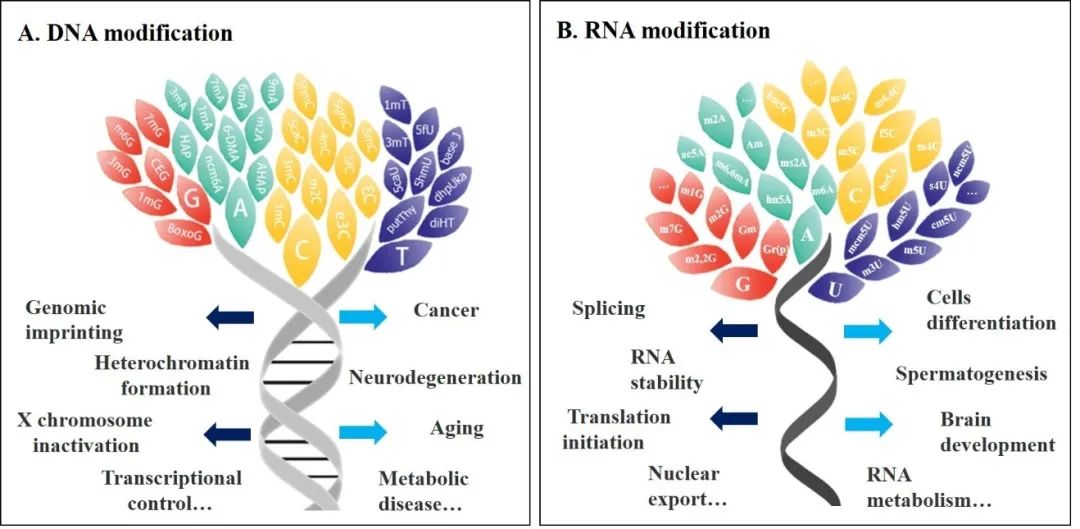
图1 核酸的各种修饰及其可能的功能。A:DNA修饰;B:RNA修饰。不同颜色的叶子代表不同类型的DNA或RNA碱基修饰。
1. 核苷酸修饰和效应蛋白
1.1 DNA 修饰
由DNA修饰介导的生物过程在过去几十年中得到了广泛的研究[16]。如图1所示,DNA的四种碱基中每一种碱基都有不同的修饰,其中5-甲基胞嘧啶(5mC)修饰是最丰富的一种。作为一种表观遗传标记,5mC与其他染色质因子紧密联系,参与多种发育和生理过程中,甚至在人类疾病中都对基因表达产生广泛的影响[17]。在哺乳动物中,负责5mC修饰添加和去除的酶已经被明确报道,其中DNMT1/3A/3B蛋白为甲基转移酶,是5mC的“编写器”,而TET1/2/3蛋白为去甲基酶,是5mC的“擦除器”[17]。这些被称为“编写器”和“擦除器”的效应蛋白,为操纵DNA的修饰状态提供了有效的工具,并可能成为潜在的治疗靶点。其他修饰如5-羟甲基胞嘧啶(5hmC)、5-甲酰基胞嘧啶(5fC)、5-羧基胞嘧啶(5caC)等虽然含量较少,但也被认为有重要的调控作用,有待进一步的研究[18]。
N6 -甲基修饰 (6mA)是另一种重要的DNA修饰,最初被认为是细菌中最普遍的DNA修饰[19,20],但是,最近研究发现,在一些真核生物中也存在这种修饰,含量要比在细菌中的修饰比例少很多[21]。在细菌中,大多数DNA甲基转移酶都是属于限制修饰(RM)系统,该系统的经典作用是防御外源核酸入侵,例如病毒入侵后会将核酸释放进入细胞,限制修饰(RM)系统会识别病毒的核酸进行切割[23]。此外,还存在一些“单独的”DNA甲基转移酶,它们可能来自祖先的限制修饰(RM)系统,但在进化过程中它们失去了的限制性内切酶搭档。其中一个典型例子是Dam甲基转移酶,它在5'-GATC-3'基序上将腺苷甲基化为6mA,相当于细菌中的writer蛋白[24]。除了Dam外,还有一大类DNA甲基转移酶作用于特定的序列基序,而对应的去甲基化酶在细菌中是不存在的[22]。
1.2 RNA 修饰
在~150种RNA修饰中,m6A是mRNA上含量最多的一种修饰,在基因表达调控中发挥重要作用,它参与了胚胎发育、神经元调节、肿瘤发生等多个生物学过程[7,25]。据估计,m6A在细胞mRNA中占总腺苷残基的0.1%-0.4%,平均每个转录本有2-3个m6A位点[10]。虽然m6A在真核生物中的存在早已经被鉴定,但是由于缺乏准确的定位方法,其具体分布情况一直不清楚,直到最近新鉴定技术的突破才确定了m6A在mRNA的分布情况[10,26,27]。随着m6A特异性抗体的发展,研究人员开始从全基因组的角度研究其分布及相关效应蛋白[28]。迄今为止, 参与m6A通路的关键调节因子在哺乳动物中都得到很好的研究和阐述,包括METTL3/14甲基化酶和相关蛋白作为 “编写器”蛋白在所需的位置添加甲基修饰,FTO / ALKBH5去甲基化酶作为“擦除器”进行删除修改,还有 YTH家族蛋白和相关的蛋白质作为“阅读器”蛋白进行解码[3]。然而,在细菌中mRNA几乎不包含修饰,只有少数的研究报告了在某些菌株中含有微量水平的mRNA修饰[29]。此外,在各项m6A研究进展的促进下,其他的修饰也开始引起人们的关注和研究兴趣。例如一个伴随m6A修饰发生的m6Am修饰,被报道会破坏mRNA的稳定,并由“编写器”蛋白PCIF1和“擦除器”蛋白FTO进行调控[30]。
2. 核酸修饰定位的方法
随着生命科学研究的深入,核酸修饰在基因表达调控中所起的作用越来越受到人们的关注。与编码核酸的调控方式不同,它有着特殊而庞大的调控机制,而解析核苷酸修饰的基因组定位则是研究其功能的关键。由于核酸修饰的种类繁多,对应的化学性质各有不同,因此每一种修饰的检测方法的也有很大差异[31]。以m6A为例,它与没有发生修饰的A碱基的组成基本一样,它不能通过普通碱基配对策略与没有修饰的A碱基区分开。因此,近年来出现了许多新方法来绘制基因组或转录组的各种修饰图谱。我们通常将这些方法归为3类,并以DNA中的5mC和RNA中的m6A为例进行描述(图2)。
策略1:抗体富集法 抗体富集法原理是利用特异性比较好的抗体对有特异性修饰的DNA或RNA片段具有较高的亲和力的特性,便于后续的免疫共沉淀进行富集从而获取修饰片段的信号。经过免疫沉淀后,富集片段(IP)比背景序列(Input)有更多的修饰,再通过高通量测序,对富集片段与背景片段的生物信息学分析,可以估计出修饰位点的“峰值”区域,在“峰值”区域内富集序列的reads明显大于背景序列的reads。这些富集方法已广泛应用于各种修饰的分析,包括DNA中的5mC (MeDIP-seq) [32] 和RNA中的m6A (MeRIP-seq)等[33]。然而,基于抗体富集的方法最大的局限性是分辨率较低,只能定位到~100 nt长度范围,而不能定位到准确的位置。更复杂的是,有些修饰是成区域分布或成簇分布才发挥功能的,而不是仅仅因为修饰的存在本身[10,27],但是,基于抗体富集的方法无法检测不同条件下的修饰密度。为了解决这些问题,研究人员进一步优化了基于抗体富集的检测方法,试图达到更好的精度,从而获取区域内甲基化的密度信息。例如,PA‐m6A‐seq通过引入外切酶策略将m6A信号的分辨率从~100 nt提高到~30 nt [34];miCLIP和m6AC-seq利用光交联策略使得逆转录过程中产生突变或发生中止来获取精确的修饰信号等。这些方法的改进都不断地在向单碱基检测精度靠近,使得方法的分辨率有进一步的提高和突破[34,35] 。
策略2:化学反应法 化学反应法是根据修饰碱基或未修饰碱基本身参与化学反应的特性进行区分的。例如,在修饰检测中运用最成功和最广泛的化学反应法是5mC的bisulfite测序(BS-seq),它的作用原理是用化学试剂重亚硫酸盐处理后,胞嘧啶(C)被转化为尿嘧啶(U),经过逆转录和PCR扩增转化,尿嘧啶(U)会被识别为胸腺嘧啶(T),但不影响5mC,经过这个方法就可以分析得到基因组中5mC的分布图谱[36,37]。另一个例子是假尿苷(Ψ)的PSI-seq,它使用CMC(N-Cyclohexyl-N′-(2-morpholinoethyl)carbodiimide)有选择地修饰假尿苷并迫使逆转录停止,由此得到Ψ精确的位置分布[38]。此外,与抗体富集策略相似,化学方法介导的免疫共沉淀也可用于修饰后的DNA或RNA片段的富集。以m7G-seq法为例,首先将m7G转化为脱碱基的位点,然后用生物素标记该位点,方便免疫共沉淀进行富集,进而分析m7G富集的模式图[39]。但是,化学反应的缺点也很明显,例如,化学反应的处理条件通常都很苛刻,容易对核酸链造成不可逆性的破坏。另外,特异性不足也是一个严重的问题,因为有些修饰碱基和正常碱基之间的差异不足以使化学试剂进行特异性的区分和反应。
策略3:酶识别反应法 酶识别反应法是利用天然的酶能识别体内某些修饰类型的特性,从而进行切割或者停止切割反应来区分修饰碱基和未修饰碱基的基序。例如,限制性内切酶属于限制修饰系统(RM系统),利用修饰作为识别内源DNA和外来入侵物的标志。利用这一特性,研究者们已经建立了基于某些修饰敏感的限制性内切酶来绘制5mC和6mA的方法[40,41]。通过对核酸内切酶的筛选,研究人员发现,细菌的毒素抗毒素(TA)系统中的MazF和ChpBK酶可以被m6A阻断,但可以切开未修饰的基序序列[42],相应地开发了m6A-REF-seq/MASTER-seq方法,实现了全转录组m6A单碱基分辨率的解析[43,44]。此外,针对5mC的检测最近开发了一种bisulfite-free的方法——EM-seq,其原理是利用TET和βGT将5 mC和5 hmC氧化成5 gmC,然后使用脱氨酶APOBEC将旁边没有发生修饰的胞嘧啶(C)脱氨基变成尿嘧啶(U)[45]。TET和βGT的酶识别方法也在TAB-seq方法中使用,用来检测基因组5hmC单碱基的分布情况[46]。还有另一种5 hmC检测方法ACE-seq,是使用脱氨酶APOBEC将正常的胞嘧啶(C)和5 mC变成尿嘧啶(U), 再通过βGT处理被保留下来5 hmC [47],将转化序列与未转化序列进行比较后,可以通过生物信息学分析得到5hmC特征。与化学方法相比,这些酶识别反应法具有更高的特异性和敏感性,但是,不是所有的修饰类型都能找到合适的酶和合适的工具蛋白进行鉴定。令人鼓舞的是,定向进化技术的发展可以帮助研究者加速这个寻找的过程,甚至可能得到活性增强的新酶,对不同的修饰都有区分能力。
值得注意的是,第三代测序技术的发展为绘制DNA和RNA修饰提供了新的发展契机,它可以在碱基读取时直接检测修饰的存在,而且适合长片段测序,对于修饰检测具有巨大的潜能。然而,由于目前三代测序的错误率高,识别能力有限,所以该方法有待于在未来的研究中得到应用[48]。
以上的这些方法各有优缺点,所以通常需要联合使用多种方法进行检测以补充各自的不足,从而取得更可靠的结果。例如,新开发bisulfite-free的方法——EM-seq综合使用了化学反应法和酶识别反应法来获取5mC和5hmC的单碱基分布信号[49]。在理想情况下,研究人员更期望只用一种简单的方法就可以在单碱基分辨率中检测出修饰后的碱基,并量化每个位点上修饰后的碱基与未修饰的碱基的比例。此外,这种方法不仅可以准确地检测出整个基因组的每一个修饰位点,而且可以尽可能少地减少样品的投入量,甚至可以实现单细胞级别的检测。要达到这些目标,我们仍然需要不断开发和创新的新的检测方法。
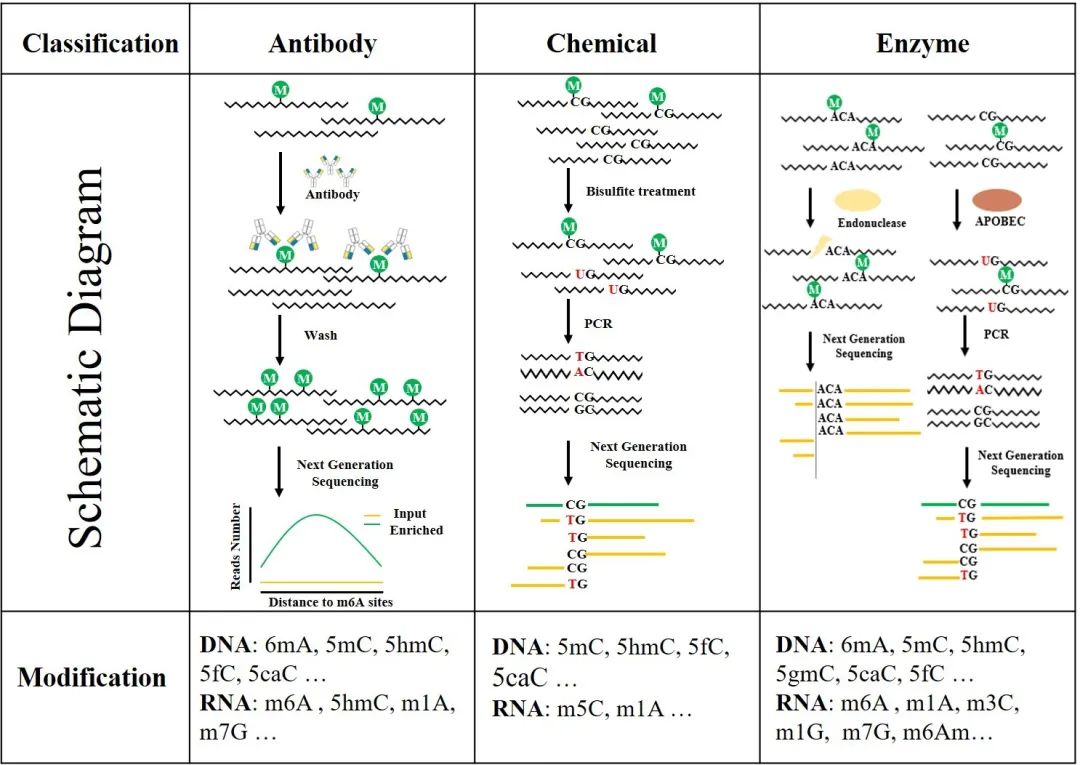
图2 核酸修饰的检测方法分类示意图。根据不同的机制,各种方法大致可分为三种:抗体富集法[28], 化学反应[36,37,50] 和酶识别反应[43,44]。修饰信号可以通过使用一种或多种策略的预处理来捕获,然后通过测序进行高通量分析。
3. 核酸修饰的编辑工具
近期研究指出,核酸修饰功能的调节是非常复杂的,有些修饰在某些位置成簇存在时才能发挥功能,而不是仅仅因为修饰的存在本身[10,27],但是我们目前许多检测工具只能实现全基因组或全转录组的检测,并不能很好评估单个碱基位点上的修饰对功能有什么作用。虽然已经有大量的研究通过干扰效应蛋白来提高或抑制整体修饰水平来说明DNA或RNA修饰的重要性,但是仍然缺乏对于特异性位点的修饰功能的直接证据。随着修饰研究的深入,相关效应蛋白如“编写”和“擦除”蛋白的逐渐被发现。在此基础上,研究人员试图将其与最新的基因编辑工具联合起来开发新的分子工具,希望通过在特定位点建立或去除修饰,从而更精确地研究这些修饰的生物学功能和相关调控机制。同时,安全有效的修饰编辑工具在临床应用中具有很大的应用前景。因此,研究者们迫切需要开发出针对特定基因组区域进行修饰状态编辑的新工具和新技术[14,51,52]。
随着CRISPR-Cas系统在基因编辑方面的进展,有几个研究小组通过改造Cas9蛋白,在癌症和胚胎干细胞等多种系统中成功地实现了DNA甲基化编辑[14,53]。Liao等人将5mC的甲基转移酶DNMT3A与dCas9(一种突变的Cas9,没有核酸内切酶活性)结合起来,在向导RNA的指导下将5mC的修饰添加到目标位点,成功地实现了DNA定向的甲基化编辑[53]。Huang等人利用DNMT3A-dCas9靶向CTCF环的在基因组上的锚定位点,并对其进行从头甲基化编辑,阻断CTCF结合DNA并干扰DNA区域性的环化,从而调控邻近区域的基因表达。为了提高编辑效率,将dCas9融合到重复抗原肽表位(Sun Tag)中,该系统可以募集多个抗体融合的DNMT3A拷贝,放大效应物的局部浓度,提高甲基化靶区的编辑效率[51]。另外, Choudhury等人通过将dCas9融合TET蛋白,实现5mC的定向氧化,而氧化产物会被内源性DNA修复机制识别并被常规胞嘧啶取代,从而实现5mC到C的编辑[54,55]。值得注意的是,DNA甲基化与其他表观遗传信号密切相关,如组蛋白修饰和染色质结构,因此编辑DNA修饰将最终重塑转录环境,并可能影响基因表达[56]。
相比于DNA来说,RNA更具有动态性和结构性,编辑的难度更大,因此为了有效地在RNA上添加或删除修饰,需要开发相应的工具进行RNA辅助定位。随着对dCas9系统的不断了解和完善,研究者们开发了一种反义寡核苷酸(PAMer)辅助dCas9蛋白来靶向RNA,并通过对DNA的类似策略来编辑相关的修饰[57,58]。Liu等人通过将dCas9与m6A “编写器”蛋白融合,开发出一种针对转录本上特定序列的分子工具,并在目标位置调节m6A修饰。通过该系统,他们发现Hsp70 mRNA的5 ' UTR上的定向修饰可以提高应激条件下的翻译效率[59]。另外,Rau K等人通过将dCas9与m6A的“擦除器”FTO融合表达,也实现了RNA导向的位点特异性去甲基化[60]。同时,本身具有RNA靶向能力的Cas13家族蛋白的发现,为RNA靶向的研究提供了新的契机。Li等人通过融合无活性的Cas13b (dCas13b)和m6A的效应蛋白,成功编辑了特异性基因的m6A修饰,并通过去甲基化调控了mRNA的稳定性(图3) [61]。
虽然CRISPR-Cas系统是目前使用最广泛靶向DNA或RNA的工具,但是还有适用于其他不同场景的可编程工具也在开发中。例如,另一种RNA引导的核酸内切酶Cpf1在基因组编辑中已被广泛应用,但在修饰编辑中的运用尚待进一步开发[62]。还有工程化的锌指结构(ZF)和转录激活子样效应子(TALEs)系统可与表观遗传效应子相连接,发挥修饰操纵作用[63]。另外, CIRTS系统(the CRISPR-Cas-inspired RNA targeting system)利用由ssRNA结合蛋白、RNA发夹结合蛋白和效应蛋白组成的工程融合蛋白,在向导RNA的辅助下靶向特定的RNA序列[64]。该工具完全是由人来源的蛋白部分进行构建的,所以在应用于疾病治疗时,可能会避免免疫排斥的问题。此外,PUF蛋白是靶向RNA的另一种选择,它可以通过取代其特殊的重复结构中的氨基酸序列,从而实现与特定的核苷酸结合[65]。
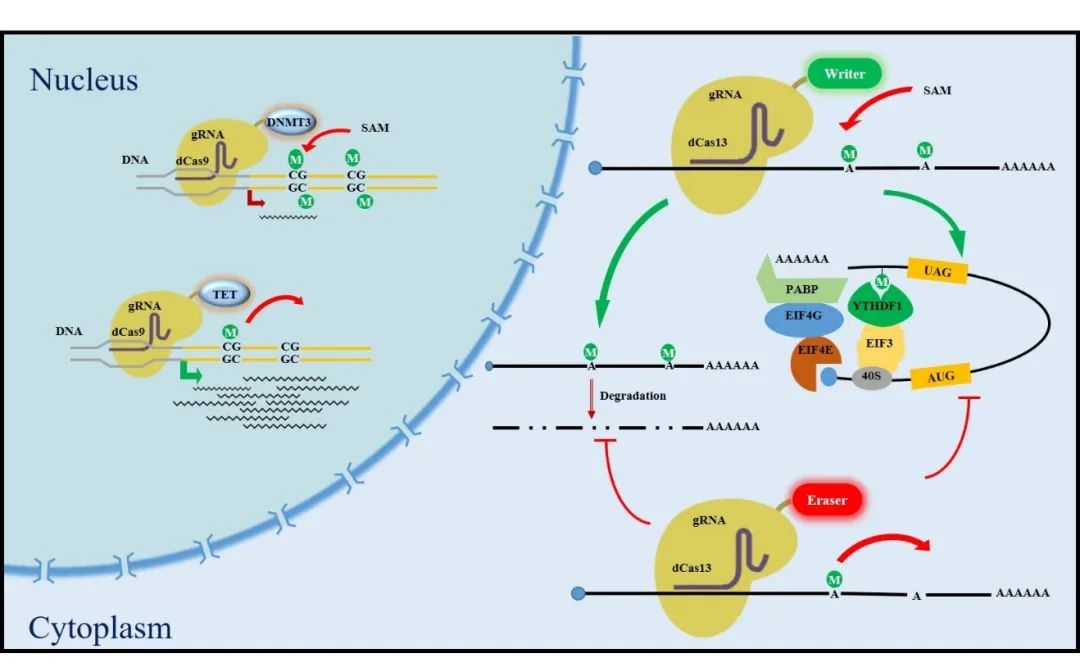
图3 基于CRISPR的DNA和RNA修饰编辑系统原理图。在细胞核内,DNA修饰5mC可通过dCas9融合效应物DNMT3 (writer)或TET (eraser)进行添加或去除,从而调控转录。在细胞质中,dCas13融合效应蛋白可添加或去除RNA上的m6A修饰,介导转录后修饰的调控,例如通过reader蛋白介导的mRNA降解或翻译抑制的调控作用。
4. 展望与不足
DNA和RNA修饰作为序列之外另一层信息的展现,彻底改变了我们对基因调控的理解,只是目前关于修饰与基因调控的因果关系仍然是一个谜。为了探究这个谜底,首先要做的就是在基因组中找到每一种修饰的确切位置。
随着新技术的发展,尤其是高通量测序的快速发展,再结合各种生物和化学方法,极大促进了各类修饰的检测体系的开发。虽然目前针对主要修饰类型的检测障碍已经不大,但是,鉴于还有上百种RNA修饰和近百种DNA修饰,在全基因组准确地将它们一一检测出来仍然是一个巨大的挑战[34,35]。而且,随着我们对修饰的认识的加深,我们逐渐需要编辑工具来改变目标区域的修饰状态,纠正疾病中错误修饰的碱基,或者在基础研究中研究特定区域不同修饰状态的相应功能,这些新的DNA和RNA编辑工具的出现能为特定基因的区域修饰机制和功能研究提供全新的角度。例如,Liu等人证明,靶向将m6A添加在Actb 基因的mRNA的3′ UTR上,会导致mRNA快速降解,而编辑相同基因mRNA的5′ UTR则没有这种效果[57]。虽然先前基于转录组生物信息学分析的研究已经提出m6A修饰的功能是区域依赖的,但这项工作首次为支持这一假设提供了直接证据。在这篇综述中,我们主要以5mC和m6A为例来介绍相关的工具和技术,因为这两种修饰在DNA和RNA中被研究的最多,相关效应蛋白被阐述地比较透彻。然而,随着各类修饰的效应蛋白逐渐被解析,上述相关方法将很方便地应用于其他修饰的检测和编辑中。
此外,这些工具的进步也为基础和临床研究提供了全新的选择。例如,利用表观遗传编辑工具对许多复杂疾病,特别是针对癌症进行了大量研究[14]。有研究构建了一个dCas9-TET1CD融合蛋白,通过去甲基化目标区域来调节肿瘤抑制基因的活性[54]。此外,这些工具还被用于探索肿瘤细胞通过失调MYC表达来劫持增强子的事件、研究通过位点特异性低甲基化上调同源框的癌基因的事件、或通过“hit-and-run”的表观遗传事件阻止衰老进入而促进肿瘤发生的机制探索等[66]。随着这些操作技术的快速发展,其应用范围已大大扩展,以探索针对更多种疾病的新型治疗策略。例如,Kantoret等人提出了一种通过dCas9-DNMT3A系统治疗帕金森病的新方法,该方法建立了SNCA第一内含子的高甲基化模式,目的是抑制其表达[67]。类似的侧重于神经退行性疾病和肌肉萎缩症的治疗研究也有报道[68]。
不可否认的是,这些工具可能也存在CRISPR系统固有的缺陷,例如脱靶效应、DNA双链断裂损伤、细胞癌化等等问题。脱靶效应是最常见的一个问题,会导致在靶点以外的位置引入意外的可遗传或表观遗传的变化[69,70]。根据最近的研究,在不同的物种中使用不同的CRISPR系统的脱靶效应是不同的[71,72]。研究者们已经在努力地开发相关方法来估计各类CRISPR系统所产生的脱靶效应,也尽可能地改进编辑工具以减少不同CRISPR系统的脱靶效应。有很多研究提出了几个可能的方法来尽可能减少脱靶效应:1、调节sgRNA-Cas9复合物的有效浓度和释放方式;2、sgRNA分子的合理设计和修饰;3、筛选新的CRISPR蛋白变体或定向改造CRISPR蛋白等[73]。随着基因组编辑技术的快速发展,基于CRISPR-Cas系统的DNA/RNA修饰编辑工具将会得到不断的完善。然而,在融合表达目的蛋白的编辑系统中,效应蛋白的引入可能会产生额外的脱靶效应。例如,在单碱基编辑器系统中,脱靶效应的主要来源不是CRISPR系统,而是效应蛋白脱氨酶导致的。如果酶的高活性导致整个基因组的过度修饰,而如果酶的低活性会使得编辑效果不佳,无法从背景中解析出预期位点的信号[74]。因此,在设计DNA/RNA修饰编辑工具之前,我们建议先仔细评估效应蛋白在不同系统中适合的工作活性,再进行下游的实验。在某些情况下,可能还需要对效应蛋白进行合理的调控,甚至是蛋白的进化来适应不同的工作系统。
综上所述,这些新的技术使我们能够更好地描述和理解核酸修饰的外观、调控机制和功能。而随着核酸修饰领域的不断快速发展,它将为基础研究和转化研究做出重要的贡献。
致谢
感谢国家自然科学基金项目(批准号: 31922015, 31622031, 91753129)和广东省自然科学基金(Grant no. 2018B030306044)项目的资助。参考文献
[1] Fu Y, He C. Nucleic acid modifications with epigenetic significance. Curr Opin Chem Biol 2012;16:516–24. https://doi.org/10.1016/j.cbpa.2012.10.002.
[2] Chen K, Zhao BS, He C. Nucleic Acid Modifications in Regulation of Gene Expression. Cell Chem Biol 2016;23:74–85. https://doi.org/10.1016/j.chembiol.2015.11.007.
[3] Shi H, Wei J, He C. Where, When, and How: Context-Dependent Functions of RNA Methylation Writers, Readers, and Erasers. Mol Cell 2019;74:640–50. https://doi.org/10.1016/j.molcel.2019.04.025.
[4] Arber W, Linn S. DNA Modification and Restriction. Annu Rev Biochem 1969;38:467–500. https://doi.org/10.1146/annurev.bi.38.070169.002343.
[5] Machnicka MA, Milanowska K, Osman Oglou O, Purta E, Kurkowska M, Olchowik A, et al. MODOMICS: a database of RNA modification pathways—2013 update. Nucleic Acids Res 2012;41:D262–7. https://doi.org/10.1093/nar/gks1007.
[6] Biswas S, Rao CM. Epigenetic tools (The Writers, The Readers and The Erasers) and their implications in cancer therapy. Eur J Pharmacol 2018. https://doi.org/10.1016/j.ejphar.2018.08.021.
[7] Barber BA, Rastegar M. Epigenetic control of Hox genes during neurogenesis, development, and disease. Ann Anat - Anat Anzeiger 2010;192:261–74. https://doi.org/10.1016/j.aanat.2010.07.009.
[8] Adli M. The CRISPR tool kit for genome editing and beyond. Nat Commun 2018;9:1911. https://doi.org/10.1038/s41467-018-04252-2.
[9] Kumar S, Chinnusamy V, Mohapatra T. Epigenetics of Modified DNA Bases: 5-Methylcytosine and Beyond. Front Genet 2018;9. https://doi.org/10.3389/fgene.2018.00640.
[10] Meyer KD, Saletore Y, Zumbo P, Elemento O, Mason CE, Jaffrey SR. Comprehensive analysis of mRNA methylation reveals enrichment in 3′ UTRs and near stop codons. Cell 2012;149:1635–46. https://doi.org/10.1016/j.cell.2012.05.003.
[11] He C. Grand Challenge Commentary: RNA epigenetics? Nat Chem Biol 2010;6:863–5. https://doi.org/10.1038/nchembio.482.
[12] Chen K, Luo GZ, He C. High-Resolution Mapping of N6-Methyladenosine in Transcriptome and Genome Using a Photo-Crosslinking-Assisted Strategy. vol. 560. 1st ed. Elsevier Inc.; 2015. https://doi.org/10.1016/bs.mie.2015.03.012.
[13] Churko JM, Mantalas GL, Snyder MP, Wu JC. Overview of High Throughput Sequencing Technologies to Elucidate Molecular Pathways in Cardiovascular Diseases. Circ Res 2013;112:1613–23. https://doi.org/10.1161/CIRCRESAHA.113.300939.
[14] Urbano, Smith, Weeks, Chatterjee. Gene-Specific Targeting of DNA Methylation in the Mammalian Genome. Cancers (Basel) 2019;11:1515. https://doi.org/10.3390/cancers11101515.
[15] Wang F, Wang L, Zou X, Duan S, Li Z, Deng Z, et al. Advances in CRISPR-Cas systems for RNA targeting, tracking and editing. Biotechnol Adv 2019;37:708–29. https://doi.org/10.1016/j.biotechadv.2019.03.016.
[16] Robertson KD. DNA methylation and human disease. Nat Rev Genet 2005;6:597–610. https://doi.org/10.1038/nrg1655.
[17] Li E, Zhang Y. DNA Methylation in Mammals. Cold Spring Harb Perspect Biol 2014;6:a019133–a019133. https://doi.org/10.1101/cshperspect.a019133.
[18] Breiling A, Lyko F. Epigenetic regulatory functions of DNA modifications: 5-methylcytosine and beyond. Epigenetics Chromatin 2015;8:24. https://doi.org/10.1186/s13072-015-0016-6.
[19] Campbell JL, Kleckner N. E. coli oriC and the dnaA gene promoter are sequestered from dam methyltransferase following the passage of the chromosomal replication fork. Cell 1990;62:967–79. https://doi.org/10.1016/0092-8674(90)90271-F.
[20] Messer W, Noyer-Weidner M. Timing and targeting: The biological functions of Dam methylation in E. coli. Cell 1988;54:735–7. https://doi.org/10.1016/S0092-8674(88)90911-7.
[21] Ratel D, Ravanat JL, Berger F, Wion D. N6-methyladenine: The other methylated base of DNA. BioEssays 2006;28:309–15. https://doi.org/10.1002/bies.20342.
[22] Casadesus J, Low D. Epigenetic Gene Regulation in the Bacterial World. Microbiol Mol Biol Rev 2006;70:830–56. https://doi.org/10.1128/MMBR.00016-06.
[23] Murray NE. Immigration control of DNA in bacteria: self versus non-self. Microbiology 2002;148:3–20. https://doi.org/10.1099/00221287-148-1-3.
[24] Wion D, Casadesús J. N6-methyl-adenine: an epigenetic signal for DNA–protein interactions. Nat Rev Microbiol 2006;4:183–92. https://doi.org/10.1038/nrmicro1350.
[25] Olynik BM, Rastegar M. The Genetic and Epigenetic Journey of Embryonic Stem Cells into Mature Neural Cells. Front Genet 2012;3. https://doi.org/10.3389/fgene.2012.00081.
[26] Linder B, Grozhik A V., Olarerin-George AO, Meydan C, Mason CE, Jaffrey SR. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nat Methods 2015;12:767–72. https://doi.org/10.1038/nmeth.3453.
[27] Dominissini D, Moshitch-Moshkovitz S, Schwartz S, Salmon-Divon M, Ungar L, Osenberg S, et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature 2012;485:201–6. https://doi.org/10.1038/nature11112.
[28] Feederle R, Schepers A. Antibodies specific for nucleic acid modifications. RNA Biol 2017;14:1089–98. https://doi.org/10.1080/15476286.2017.1295905.
[29] Mohanty BK, Kushner SR. The majority of Escherichia coli mRNAs undergo post-transcriptional modification in exponentially growing cells. Nucleic Acids Res 2006;34:5695–704. https://doi.org/10.1093/nar/gkl684.
[30] Akichika S, Hirano S, Shichino Y, Suzuki T, Nishimasu H, Ishitani R, et al. Cap-specific terminal N 6 -methylation of RNA by an RNA polymerase II–associated methyltransferase. Science 2019;363:eaav0080. https://doi.org/10.1126/science.aav0080.
[31] Mongan NP, Emes RD, Archer N. Detection and analysis of RNA methylation. F1000Research 2019;8:559. https://doi.org/10.12688/f1000research.17956.1.
[32] Mohn F, Weber M, Schübeler D, Roloff T-C. Methylated DNA Immunoprecipitation (MeDIP), 2009, p. 55–64. https://doi.org/10.1007/978-1-59745-522-0_5.
[33] Dominissini D, Moshitch-Moshkovitz S, Salmon-Divon M, Amariglio N, Rechavi G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nat Protoc 2013;8:176–89. https://doi.org/10.1038/nprot.2012.148.
[34] Chen K, Lu Z, Wang X, Fu Y, Luo G-Z, Liu N, et al. High-Resolution N 6 -Methyladenosine (m 6 A) Map Using Photo-Crosslinking-Assisted m 6 A Sequencing. Angew Chemie Int Ed 2015;54:1587–90. https://doi.org/10.1002/anie.201410647.
[35] Fu Y, Luo GZ, Chen K, Deng X, Yu M, Han D, et al. N6-methyldeoxyadenosine marks active transcription start sites in Chlamydomonas. Cell 2015;161:879–92. https://doi.org/10.1016/j.cell.2015.04.010.
[36] Frommer M, McDonald LE, Millar DS, Collis CM, Watt F, Grigg GW, et al. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc Natl Acad Sci 1992;89:1827–31. https://doi.org/10.1073/pnas.89.5.1827.
[37] Li Y, Tollefsbol TO. DNA Methylation Detection: Bisulfite Genomic Sequencing Analysis, 2011, p. 11–21. https://doi.org/10.1007/978-1-61779-316-5_2.
[38] Lovejoy AF, Riordan DP, Brown PO. Transcriptome-Wide Mapping of Pseudouridines: Pseudouridine Synthases Modify Specific mRNAs in S. cerevisiae. PLoS One 2014;9:e110799. https://doi.org/10.1371/journal.pone.0110799.
[39] Enroth C, Poulsen LD, Iversen S, Kirpekar F, Albrechtsen A, Vinther J. Detection of internal N7-methylguanosine (m7G) RNA modifications by mutational profiling sequencing. Nucleic Acids Res 2019;47:e126–e126. https://doi.org/10.1093/nar/gkz736.
[40] Jin S-G, Kadam S, Pfeifer GP. Examination of the specificity of DNA methylation profiling techniques towards 5-methylcytosine and 5-hydroxymethylcytosine. Nucleic Acids Res 2010;38:e125–e125. https://doi.org/10.1093/nar/gkq223.
[41] Luo G-Z, Wang F, Weng X, Chen K, Hao Z, Yu M, et al. Characterization of eukaryotic DNA N6-methyladenine by a highly sensitive restriction enzyme-assisted sequencing. Nat Commun 2016;7:11301. https://doi.org/10.1038/ncomms11301.
[42] Imanishi M, Tsuji S, Suda A, Futaki S. Detection of N 6 -methyladenosine based on the methyl-sensitivity of MazF RNA endonuclease. Chem Commun 2017;53:12930–3. https://doi.org/10.1039/C7CC07699A.
[43] Badian NA. Reading disability in an epidemiological context incidence and environmental correlates. J Learn Disabil 1984;17:129–36. https://doi.org/10.1177/002221948401700301.
[44] Garcia-Campos MA, Edelheit S, Toth U, Safra M, Shachar R, Viukov S, et al. Deciphering the “m6A Code” via Antibody-Independent Quantitative Profiling. Cell 2019:1–17. https://doi.org/10.1016/j.cell.2019.06.013.
[45] Vaisvila R. EM-seq: Detection of DNA Methylation at Single Base Resolution from Picograms of DNA. J Chem Inf Model 2019;53:1689–99. https://doi.org/10.1017/CBO9781107415324.004.
[46] Yu M, Han D, Hon GC, He C. Tet-Assisted Bisulfite Sequencing (TAB-seq), 2018, p. 645–63. https://doi.org/10.1007/978-1-4939-7481-8_33.
[47] Schutsky EK, Denizio JE, Hu P, Liu MY, Nabel CS, Fabyanic EB, et al. Nondestructive, base-resolution sequencing of 5-hydroxymethylcytosine using a DNA deaminase. Nat Biotechnol 2018;36:1083–90. https://doi.org/10.1038/nbt.4204.
[48] Ardui S, Ameur A, Vermeesch JR, Hestand MS. Single molecule real-time (SMRT) sequencing comes of age: applications and utilities for medical diagnostics. Nucleic Acids Res 2018;46:2159–68. https://doi.org/10.1093/nar/gky066.
[49] Liu Y, Siejka-Zielińska P, Velikova G, Bi Y, Yuan F, Tomkova M, et al. Bisulfite-free direct detection of 5-methylcytosine and 5-hydroxymethylcytosine at base resolution. Nat Biotechnol 2019;37:424–9. https://doi.org/10.1038/s41587-019-0041-2.
[50] Boschi-Muller S, Motorin Y. Chemistry enters nucleic acids biology: Enzymatic mechanisms of RNA modification. Biochem 2013;78:1392–404. https://doi.org/10.1134/S0006297913130026.
[51] Huang Y-H, Su J, Lei Y, Brunetti L, Gundry MC, Zhang X, et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biol 2017;18:176. https://doi.org/10.1186/s13059-017-1306-z.
[52] Molla KA, Yang Y. CRISPR/Cas-Mediated Base Editing: Technical Considerations and Practical Applications. Trends Biotechnol 2019;xx. https://doi.org/10.1016/j.tibtech.2019.03.008.
[53] Liao J, Karnik R, Gu H, Ziller MJ, Clement K, Tsankov AM, et al. Targeted disruption of DNMT1, DNMT3A and DNMT3B in human embryonic stem cells. Nat Genet 2015;47:469–78. https://doi.org/10.1038/ng.3258.
[54] Choudhury SR, Cui Y, Lubecka K, Stefanska B, Irudayaraj J. CRISPR-dCas9 mediated TET1 targeting for selective DNA demethylation at BRCA1 promoter. Oncotarget 2016;7. https://doi.org/10.18632/oncotarget.10234.
[55] Bhutani N, Burns DM, Blau HM. DNA Demethylation Dynamics. Cell 2011;146:866–72. https://doi.org/10.1016/j.cell.2011.08.042.
[56] Pillemer E, Whitlock C, Weissman IL. Transformation-associated proteins in murine B-cell lymphomas that are distinct from Abelson virus gene products. Proc Natl Acad Sci U S A 1984;81:4434–8. https://doi.org/10.1073/pnas.81.14.4434.
[57] Liu X-M, Zhou J, Mao Y, Ji Q, Qian S-B. Programmable RNA N6-methyladenosine editing by CRISPR-Cas9 conjugates. Nat Chem Biol 2019. https://doi.org/10.1038/s41589-019-0327-1.
[58] Wei J, He C. Site-specific m6A editing. Nat Chem Biol 2019;15:848–9. https://doi.org/10.1038/s41589-019-0349-8.
[59] Tsuchida Y, Kaneko Y, Kanda N, Makino S, Utakoji T, Saito S. Possible relationship of chromosome abnormalities and gene amplification with effects of chemotherapy: a neuroblastoma xenograft study. Prog Clin Biol Res 1985;175:171–80.
[60] Rau K, Rösner L, Rentmeister A. Sequence-specific m 6 A demethylation in RNA by FTO fused to RCas9. RNA 2019;25:1311–23. https://doi.org/10.1261/rna.070706.119.
[61] Li J, Chen Z, Chen F, Ling Y, Peng Y, Luo N, et al. Targeted mRNA demethylation using an engineered dCas13b-ALKBH5 fusion protein. BioRxiv 2019:614859. https://doi.org/10.1101/614859.
[62] Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, et al. Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System. Cell 2015. https://doi.org/10.1016/j.cell.2015.09.038.
[63] Konermann S, Brigham MD, Trevino AE, Hsu PD, Heidenreich M, Le Cong, et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature 2013;500:472–6. https://doi.org/10.1038/nature12466.
[64] Rauch S, He E, Srienc M, Zhou H, Zhang Z, Dickinson BC. Programmable RNA-Guided RNA Effector Proteins Built from Human Parts. Cell 2019;178:122-134.e12. https://doi.org/10.1016/j.cell.2019.05.049.
[65] Shinoda K, Suda A, Otonari K, Futaki S, Imanishi M. Programmable RNA methylation and demethylation using PUF RNA binding proteins. Chem Commun 2020;56:1365–8. https://doi.org/10.1039/C9CC09298F.
[66] Schuijers J, Manteiga JC, Weintraub AS, Day DS, Zamudio AV, Hnisz D, et al. Transcriptional Dysregulation of MYC Reveals Common Enhancer-Docking Mechanism. Cell Rep 2018;23:349–60. https://doi.org/10.1016/j.celrep.2018.03.056.
[67] Kantor B, Tagliafierro L, Gu J, Zamora ME, Ilich E, Grenier C, et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Mol Ther 2018;26:2638–49. https://doi.org/10.1016/j.ymthe.2018.08.019.
[68] Young AB. Four Decades of Neurodegenerative Disease Research: How Far We Have Come! J Neurosci 2009;29:12722–8. https://doi.org/10.1523/JNEUROSCI.3767-09.2009.
[69] Kim D, Bae S, Park J, Kim E, Kim S, Yu HR, et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods 2015;12:237–43. https://doi.org/10.1038/nmeth.3284.
[70] Zhang X-H, Tee LY, Wang X-G, Huang Q-S, Yang S-H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Mol Ther - Nucleic Acids 2015;4:e264. https://doi.org/10.1038/mtna.2015.37.
[71] Li J, Manghwar H, Sun L, Wang P, Wang G, Sheng H, et al. Whole genome sequencing reveals rare off‐target mutations and considerable inherent genetic or/and somaclonal variations in CRISPR Cas9‐edited cotton plants. Plant Biotechnol J 2019;17:858–68. https://doi.org/10.1111/pbi.13020.
[72] Fu Y, Foden JA, Khayter C, Maeder ML, Reyon D, Joung JK, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol 2013;31:822–6. https://doi.org/10.1038/nbt.2623.
[73] Tycko J, Myer VE, Hsu PD. Methods for Optimizing CRISPR-Cas9 Genome Editing Specificity. Mol Cell 2016;63:355–70. https://doi.org/10.1016/j.molcel.2016.07.004.
[74] Jin S, Zong Y, Gao Q, Zhu Z, Wang Y, Qin P, et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science 2019:eaaw7166. https://doi.org/10.1126/science.aaw7166.
扫码关注我们
生物信息与表观组学
微信号:luolab2017
原文链接(点击下方“阅读原文”即可跳转)
https://doi.org/10.1016/j.csbj.2020.03.010
勤能补拙,过目不忘,提高m6A助力好记性?中科院王秀杰/杨运桂合作最新成果
中大骆观正等在Cell Research发文阐述肠道菌群和m6A的关系
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集