高级转录组分析和R语言数据可视化课程全部转为视频课
常规转录组是我们最常接触到的一种高通量测序数据类型,其实验方法成熟,花费较低,是大部分CNS必备的技术,现在就如做个PCR一样常见。而且分析思路简洁清晰,是入门生信,学习生信分析思路和数据可视化的首选。
数据分析是相通的,通过一个简单的课程理解其中的原理,就可以推而广之,延伸到其它类型的数据分析,如扩增子、宏基因组、单细胞等。
高级转录组分析和R数据可视化在线下举办了11期,目前因疫情停掉了线下课程,全部转为视频课,并定期提供群内文字指导和群视频指导。本课程系统讲述基于和不基于比对的转录组分析流程,从原始数据到表达矩阵、差异基因(批次处理)、可变剪接、富集分析、加权共表达网络、通路分析、可视化绘图等一系列常见操作,理论和实践兼备。
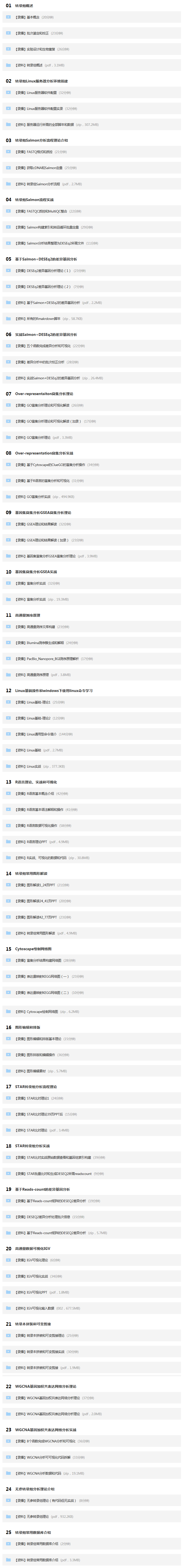
课程大纲
该课程整合了线下举办的11期内容,整个过程都比较成熟,可以在最短时间学习最多知识。后续也根据学员学习过程中提出的问题增录新的视频章节,继续完善更新。点击阅读原文跳转课程页面。
往期评价

教程内容简介如下:
转录组的应用、设计和案例分享
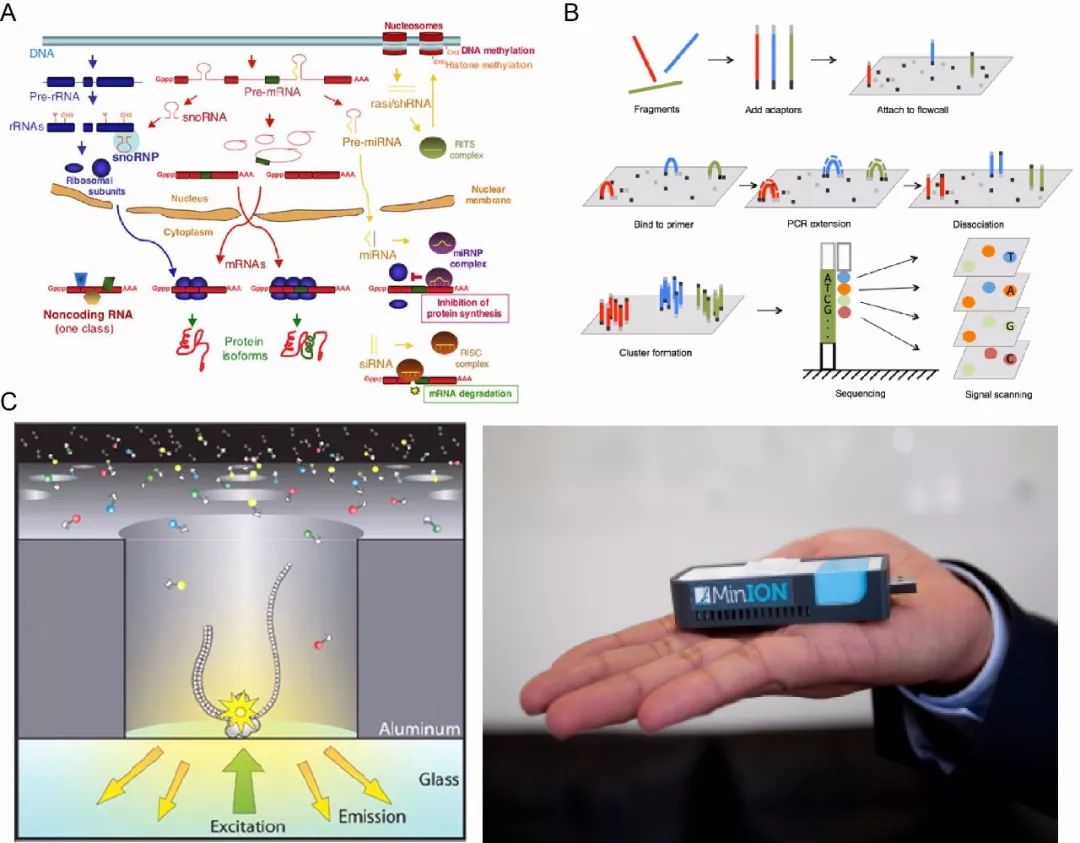
转录组学研究技术介绍
转录组学实验设计和测序原则、注意事项
二代、三代测序过程和原理解析
转录组学文章案例分析
在线基因表达资源数据库
转录组分析流程实战
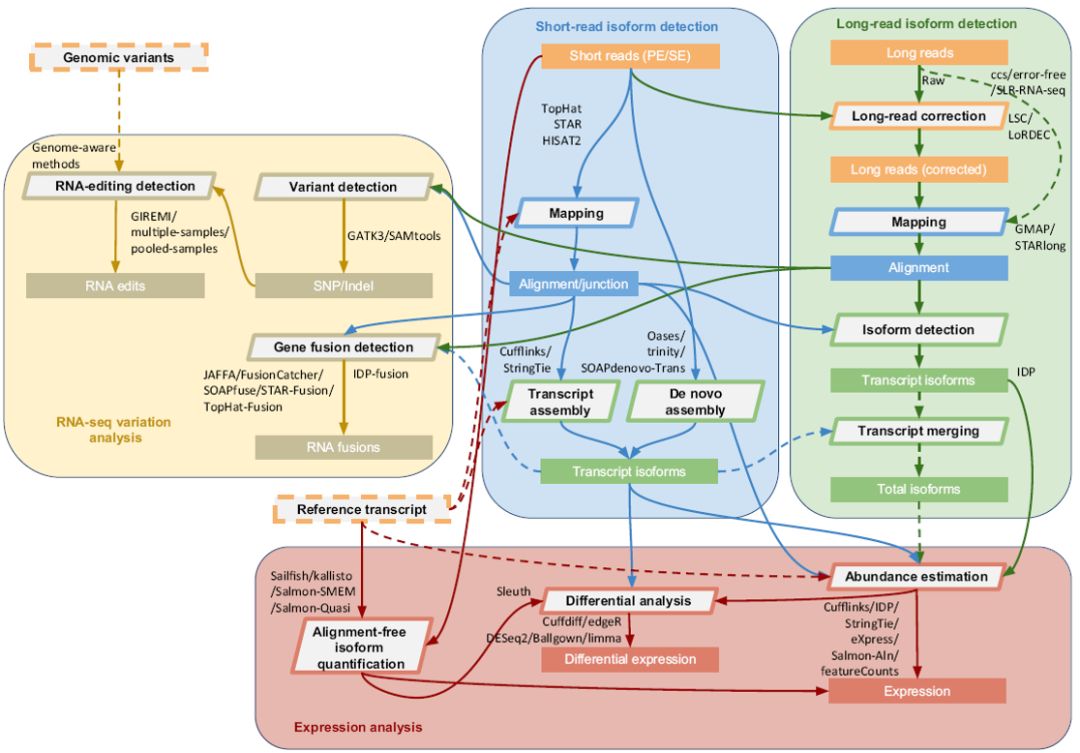
不基于比对的差异基因分析
基于比对的差异基因分析
转录本组装和选择性剪接分析
转录组高级分析
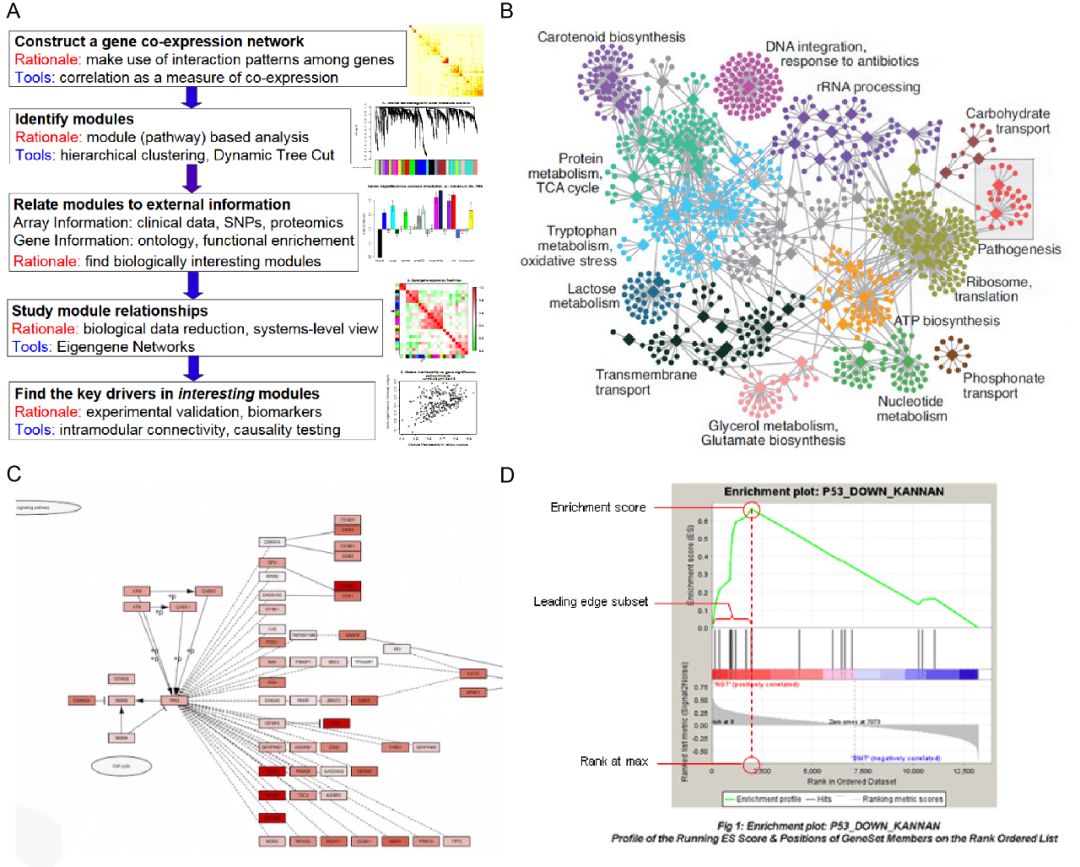
WGCNA基因、表型关联分析
转录组常见图形在线绘制
KEGG/Reactome通路图绘制,表达映射
常见图表解读和图形编辑排版
在培训上,结合发表高水平文章,进一步讲解16种常用分析图的原理和使用范围,让你不仅读懂图,更知道如何应用于自己的研究,并亲自轻松完成绘图。
针对大家使用R语言绘图学习时间成本较高的问题,易生信团队针对常用16种图开发了免费绘图网站,一键出图,更可鼠标点选参数修改图形的个性样式。
成果发表是科研过程中不可缺的一部分,发表成果又少不了图形展示。文章图表排版是否整齐规范、协调一致、重点突出对一篇文章的发表也是有不少贡献的。之前推出的文章发表图的修改和排版讲演了部分图形编辑和排版操作,本次培训也会实践从原始图形、到细节修饰再到排版发表的整个过程和注意事项。基因组浏览器用于多组学数据的可视化和关联分析,本地有IGV,在线有UCSC genome Browser和Epigenomebrowser,各有特色。
生信基础知识
Linux/Windows下Rstudio和Linux命令的使用
Linux/Windows下转录组分析流程的搭建
(如果基础薄弱,报名付款成功后,可免费领取基础程序课,做好准备工作, 让程序成为我们的得力工具而不是学习新知识的绊脚石。)
定制内容
如果您看到文章中有哪些图或分析工作需要重现,也请提出,一起讲述。
如果您有其它关注的问题,也请报名时提出,把这次课程变成您的定制讲解。
易生信系列培训课程,扫码获取免费资料
更多阅读
后台回复“生信宝典福利第一波”获取教程合集