AlphaFold 2再发350000种蛋白质结构预测结果,解锁98.5%人类蛋白质组!
共 3712字,需浏览 8分钟
·
2021-07-26 21:09
新智元报道
新智元报道
来源:DeepMind
编辑:Priscilla Emil
【新智元导读】蛋白质结构预测「资深玩家」DeepMind又发大招,时隔一周再次在nautre发文:AlphaFold已经预测出了350000种蛋白质结构,涵盖了几乎所有人类蛋白质组以及20种生物的蛋白质,现可通过公布的数据库查看各个蛋白质的3D结构。
上周在科学界发生了十分有戏剧性的一幕:两大科学顶刊Science和nature同时发文,两支研究团队都表示可以通过AI模型预测蛋白质和一些分子复合物的精确3D原子结构。
来自华盛顿大学的科研团队在Science上公布了RoseTTAFold,宣称这个模型的性能与AlphaFold 2相当。
并且其端到端版本在RTX2080上生成少于400个残基的蛋白质的骨干坐标仅需10分钟。
 RoseTTAFold蛋白质预测工具的论文
RoseTTAFold蛋白质预测工具的论文
这显然让谷歌DeepMind团队有些措手不及,匆匆忙忙选择在nature上开源了AlphaFold 2的技术细节。
不过今天故事似乎要反转:DeepMind团队在nature上再接再厉,公布了他们已经使用AlphaFold工具预测的来自人类和20种其他生物共350000种蛋白质的结构,例如大肠杆菌、酵母菌和果蝇等等。

论文链接:https://www.nature.com/articles/s41586-021-03828-1
目前他们已经复现了44%的人类蛋白质结构,覆盖了人类基因组编码的所有氨基酸结构位置的近60%。
不仅如此,这个团队将在未来的几个月里,通过计算预测目前UniRef目录中所有科学界已知的,经过测序的1亿个蛋白质分子结构。
而AlphaFold与EMBL-EBI(欧洲分子生物学实验室)合作推出了蛋白质结构数据库(链接:https://alphafold.ebi.ac.uk/),科研人员只要输入蛋白质名称或编号,就可以迅速获得蛋白质的3D结构图像。

模型好不好,还得看疗效。看来AlphaFold这一次靠「钞能力」赢了。
AlphaFold新进展
AlphaFold新进展
昨日,DeepMind在nature上刊登的文章,描述了AlphaFold对人类蛋白质组的结构预测,覆盖了98.5%的所有人类蛋白质组,还对20种其它生物蛋白质的结构进行了预测。
这20种其它生物包括大肠杆菌、酵母菌、果蝇等,它们都是生物研究的重点。
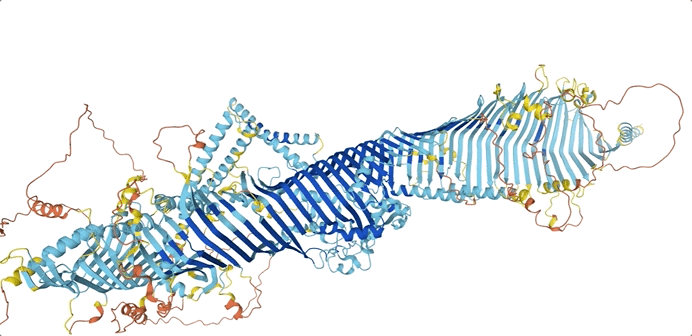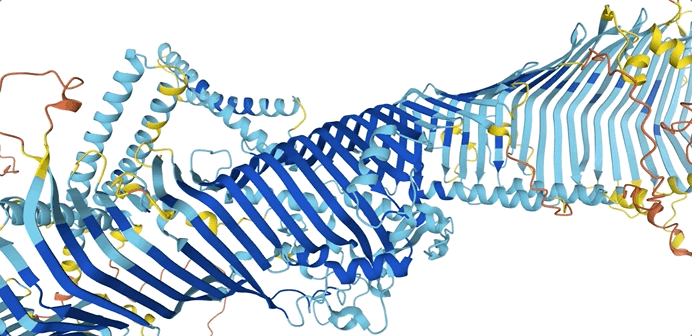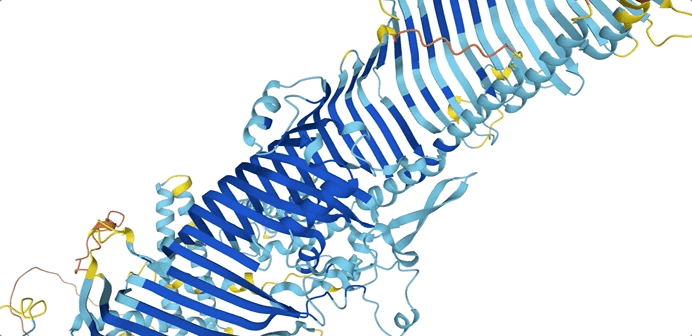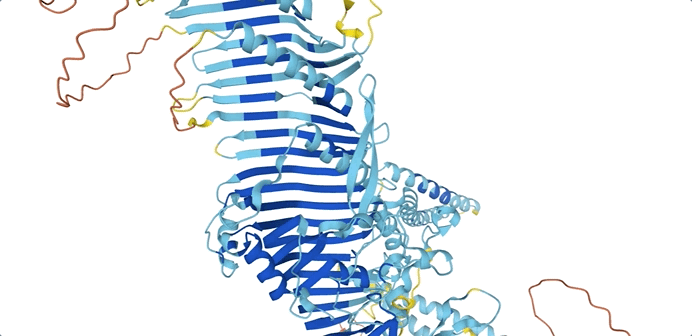
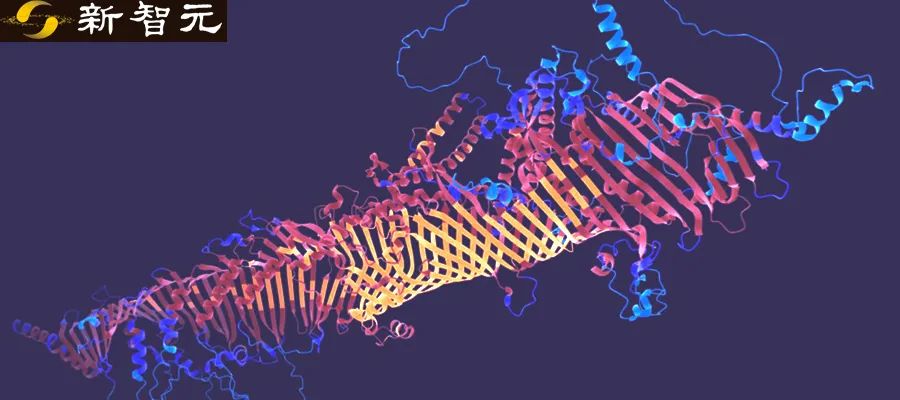
果蝇蛋白质3D结构示意图
在论文中,作者表示,AlphaFold能够对人类蛋白质组35.7%氨基酸结构位置进行预测,且有很高的置信度,是实验方法覆盖的结构数量的两倍。
在单个蛋白质水平上,AlphaFold能够对43.8%的蛋白中,至少四分之三的氨基酸序列给出了可信预测。

在单个蛋白质的介绍中,数据库列出了蛋白质的基本信息,包括蛋白质名称、基因、来源生物、生物功能等。

以T细胞免疫调节蛋白同源物为例,这个蛋白质可以通过免疫调节保护寄生虫免受宿主免疫系统的攻击。
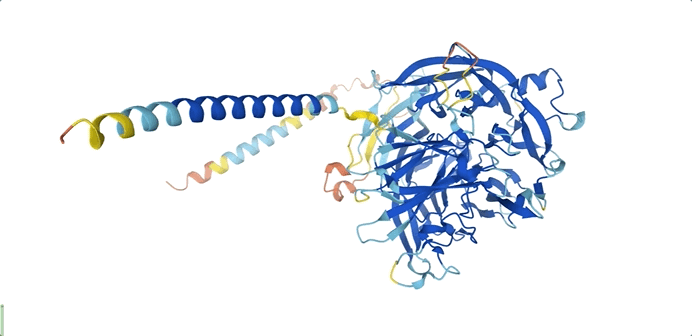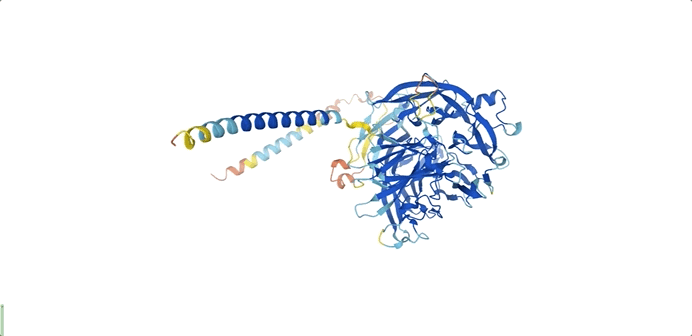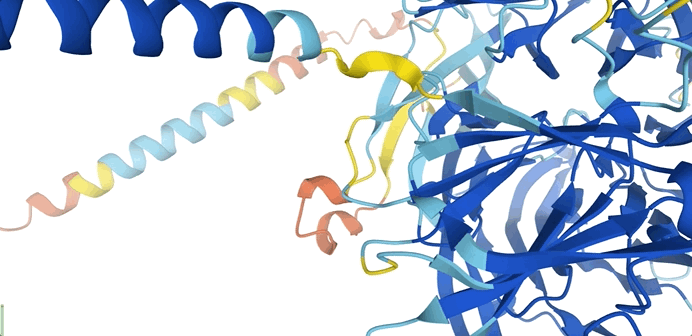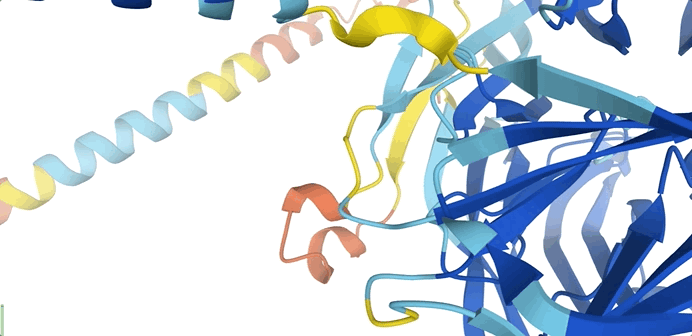
恶性疟原虫T细胞免疫调节蛋白同源物结构图
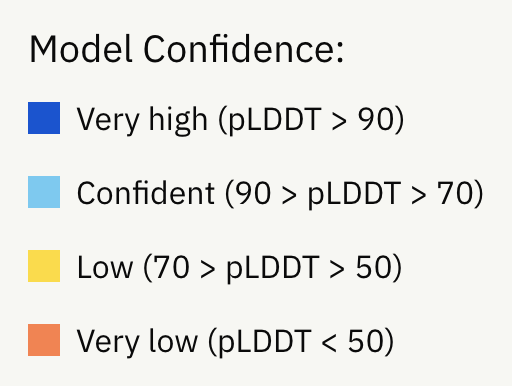
如上图所示,DeepMind在数据库公布的蛋白质结构预测模型中,不同的颜色表示不同的预测可信度。
残基置信度分数(pLDDT)范围在0-100,数值大于90指信度非常高,表示为蓝色;数值为70-90表示为浅蓝色;数值为50-70表示为黄色;橙色部分表示信度非常低,数值小于50,有可能是孤立的非结构化区域。
除了公布蛋白质基本信息及模型结构外,网站还公布了蛋白质结构的预测对齐误差,后续还会推出蛋白质序列特征功能。
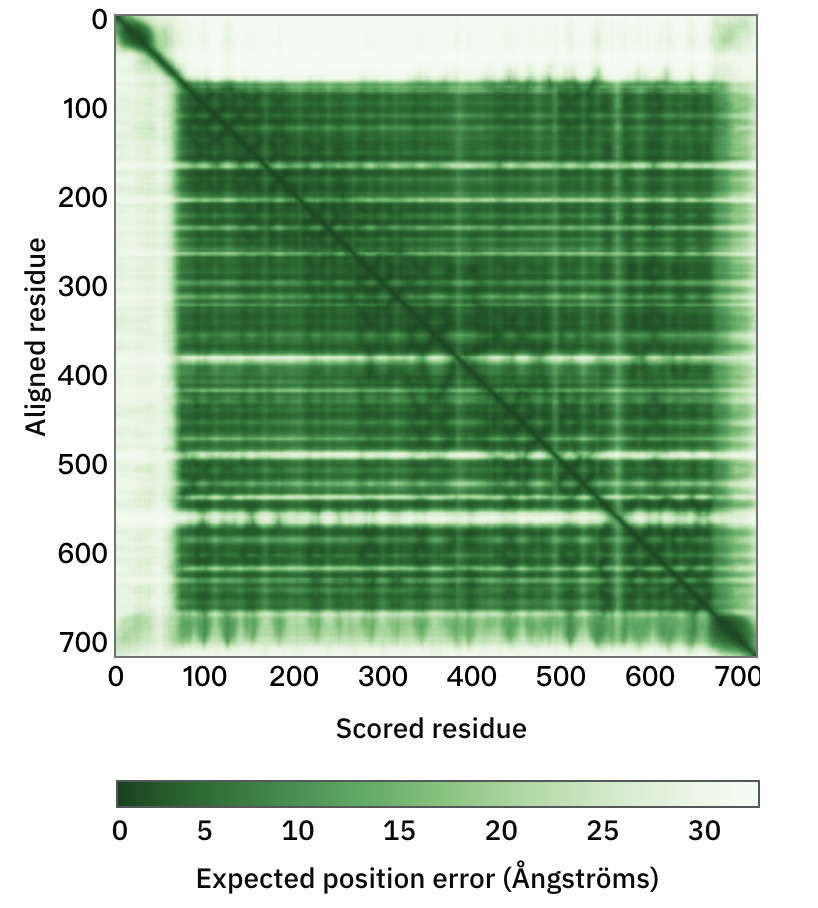
蛋白质结构模型的预测对齐误差
AlphaFold 2预测出了约20000种人类基因组蛋白质,加上20种其它生物的蛋白质组的结构预测,总计公布了超过350000种蛋白质结构预测模型。
「AlphaFold确定了覆盖几乎整个人类蛋白质组(98.5%的所有人类蛋白,即Uniprot里所有2700aa以下的蛋白)的蛋白质的结构。」
DeepMind表示,接下来的几个月将计划扩大蛋白质模型列表,以涵盖所有编目的蛋白质,共计大约1亿个分子。
AlphaFold为近44%的人类蛋白质预测了结构,涵盖了人类基因组中60%的氨基酸结构位置预测。
AlphaFold确定了许多其他人类蛋白质是「无序的」,也就是说,蛋白质的形状不采用单一结构。
当这种无序的蛋白质与另一蛋白质结合时,最终可能会确定出一种结构。
当然,这些蛋白质也可能会有多种结构。
蛋白质结构预测:50年的难题
蛋白质结构预测:50年的难题
蛋白质的 3D 结构在很大程度上决定了它的功能,通过研究蛋白质的3D结构,生物学家不仅可以更加快速地研发出针对各类疾病的药物,甚至能够揭开生命之谜。
弄清楚蛋白质折叠成什么形状被称为“蛋白质折叠问题”,在过去的50年里一直是生物学的一个巨大挑战。
通过结构测定是一个有效但非常费力的方法,它比基因组测定难度大得多。
按照传统的实验步骤,从基因序列到相应的蛋白质结构测定之间还要经过基因表达、蛋白质的提取和纯化、结晶、X射线衍射分析等步骤。由于蛋白质结构和性质的多样性,这些步骤大多没有固定的规律可循。
并且试验需要反复进行多次,每个结构的测定可能都需要数年的工作,并且动用价值数百万元的设备。

在1972年诺贝尔化学奖的获奖感言中,克里斯蒂安·安芬森( Christian Anfinsen ) 提出了一个著名假设:理论上,蛋白质的氨基酸序列应该完全决定其结构。

但是从理论上分析,蛋白质在形成之前,可以折叠出天文数字的3D结构。
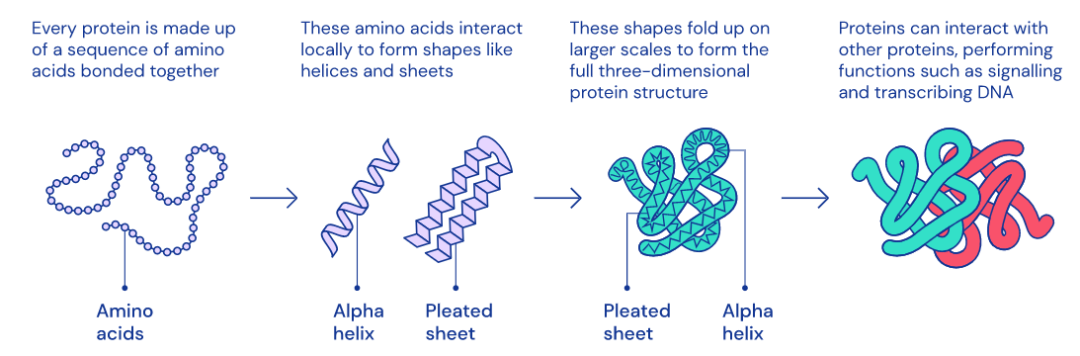
1969年,赛勒斯·莱文塔尔 (Cyrus Levinthal) 指出,通过蛮力计算列举出典型蛋白质的所有可能构型所需的时间比已知宇宙的年龄还要长。

他预测,一个典型的蛋白质可能有10的300次方种折叠形式。
如今DeepMind的团队用AI解开了这道世纪难题。
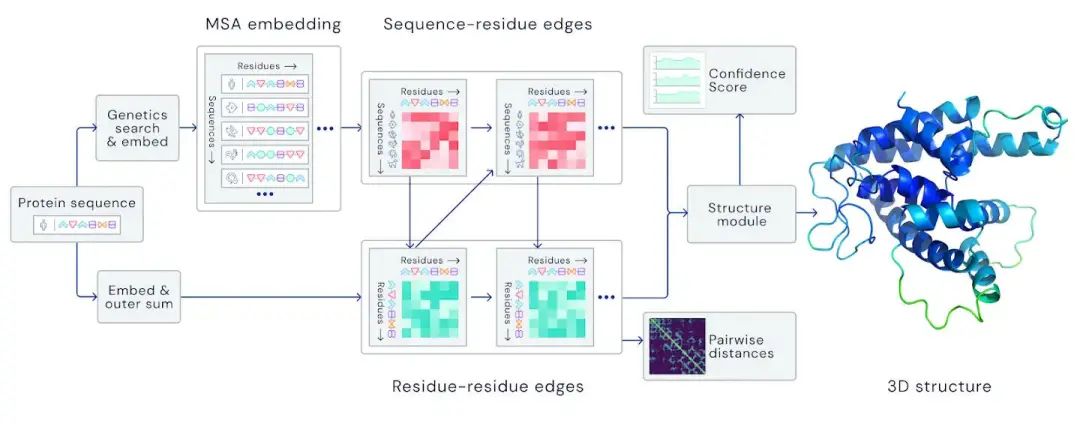
经过训练的深度神经网络可以根据蛋白质的基因序列预测蛋白质的特性。主要判断依据是氨基酸对之间的距离和连接这些氨基酸的化学键之间的角度。
通过已知信息,可以推断出蛋白质折叠之后的角度和距离信息,从而推断出整个蛋白质的结构。
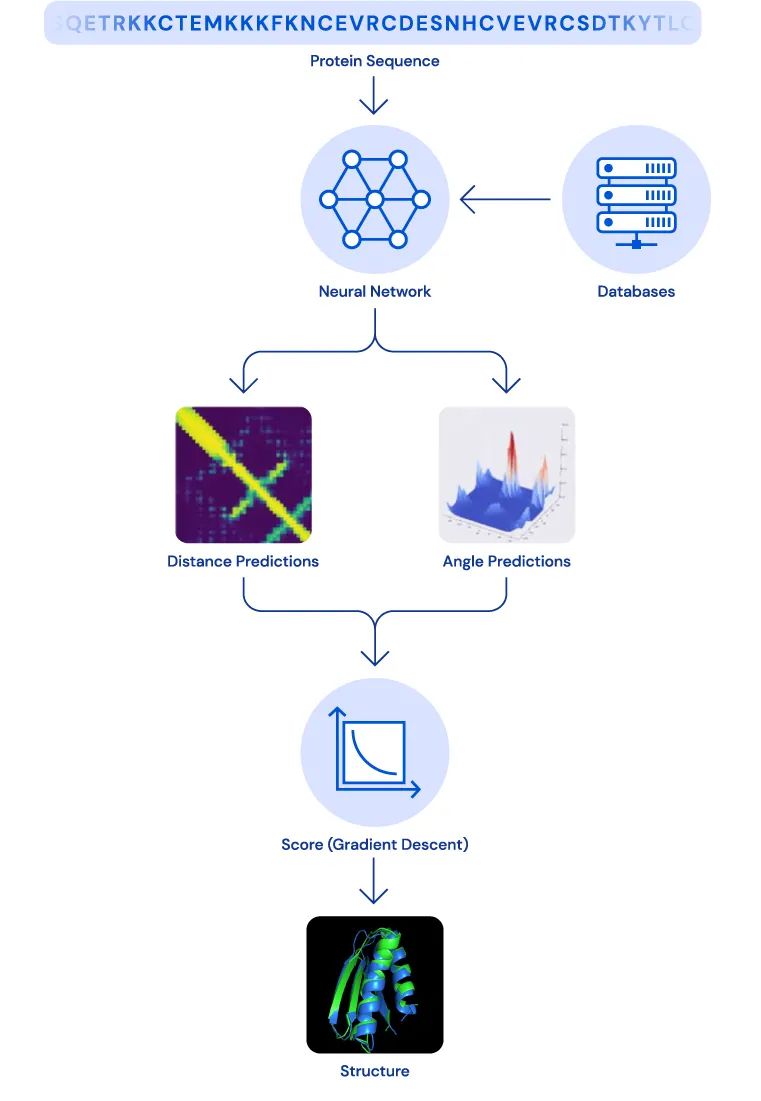
AlphaFold数据库的建立,把生物科学家们从研究蛋白质时最费时费力的结构测定工作中解脱了出来,从而可以专心转向研究的核心部分。
「我们原来花费数个月甚至数年时间才完成的事情,AlphaFold在一个周末就能完成」,朴茨茅斯大学结构生物学教授兼酶创新中心 (CEI) 中心主任感慨道。
在AlphaFold的帮助下,朴茨茅斯大学酶创新中心正在设计一种酶,来加速降解一次性塑料。
科罗拉多大学博尔德分校的一个团队在使用AlphaFold来预测研究抗生素耐药性的前景。
更多的可能性还在不断被发掘。
业内不只有AlphaFold
业内不只有AlphaFold
除了AlphaFold和RoseTTAFold两大工具以外,商业上也有与蛋白质预测相关的消息。
成立于2011年,专注于在大肠杆菌中制造蛋白质的公司Absci在周四上市。

今年1月,Absci收购了Denovium,一家使用深度学习人工智能对蛋白质进行分类和预测的公司,目前已经接受了1亿种蛋白质的训练。
Absci在6月还收购了Totient,一家分析免疫系统对某些疾病的反应的生物技术公司。
这样一来,Absci就具备了蛋白质制造、评估和筛选能力,完善了从蛋白质制造和测试到人工智能支持的药物开发。
随着人工智能的不断发展,人工智能有可能彻底改变21世纪的科学工作方式。
人工智能对蛋白质大规模的准确结构预测,将成为推动生物医学界发展的重要工具,从蛋白质的结构解决新的科学问题。
热切期待AlphaFold继续帮助科学界解锁下一步的发现。
参考资料:
https://deepmind.com/blog/article/putting-the-power-of-alphafold-into-the-worlds-hands
https://www.nature.com/articles/s41586-021-03828-1_reference.pdf

