哈佛大学单细胞课程|笔记汇总 (一)
生物信息学习的正确姿势
NGS系列文章包括NGS基础、转录组分析 (Nature重磅综述|关于RNA-seq你想知道的全在这)、ChIP-seq分析 (ChIP-seq基本分析流程)、单细胞测序分析 (重磅综述:三万字长文读懂单细胞RNA测序分析的最佳实践教程 (原理、代码和评述))、DNA甲基化分析、重测序分析、GEO数据挖掘(典型医学设计实验GEO数据分析 (step-by-step) - Limma差异分析、火山图、功能富集)等内容。
哈佛大学单细胞课程|笔记汇总
为什么做单细胞?
如何得到单细胞原始数据并转换成分析需要的矩阵格式
质控前的数据准备
质控
归一化和主成分分析
聚类分析与可视化
marker识别与注释
哈佛大学单细胞课程|笔记汇总
单细胞转录组测序进展迅速,伴随而来的是许许多多的内容与讲义,很多课程都很长。。。嗨像我这样莫有耐心的小孩子,能完整看完的只有一个 —— Single-cell RNA-seq analysis workshop(https://github.com/hbctraining/scRNA-seq)。我将把教程内容尽量汇总,你需要做的就是点击收藏慢慢看!
撰稿人:协和 张虎
(一)Why single-cell RNA-seq
在整个人体组织中,细胞类型、状态和相互作用非常多样。为了更好地了解这些组织存在的细胞类型,单细胞RNA-seq(scRNA-seq)提供了在单细胞水平上表达基因的信息。
单细胞转录组测序可用于 :
探索组织中存在哪些细胞类型
识别未知/稀有的细胞类型或状态
阐明分化过程中或跨时间或状态的基因表达变化
识别在不同条件(例如治疗或疾病)下特定细胞类型中差异表达的基因
探索细胞类型之间的表达变化,同时纳入空间、调控和/或蛋白质信息
常见方法包括:
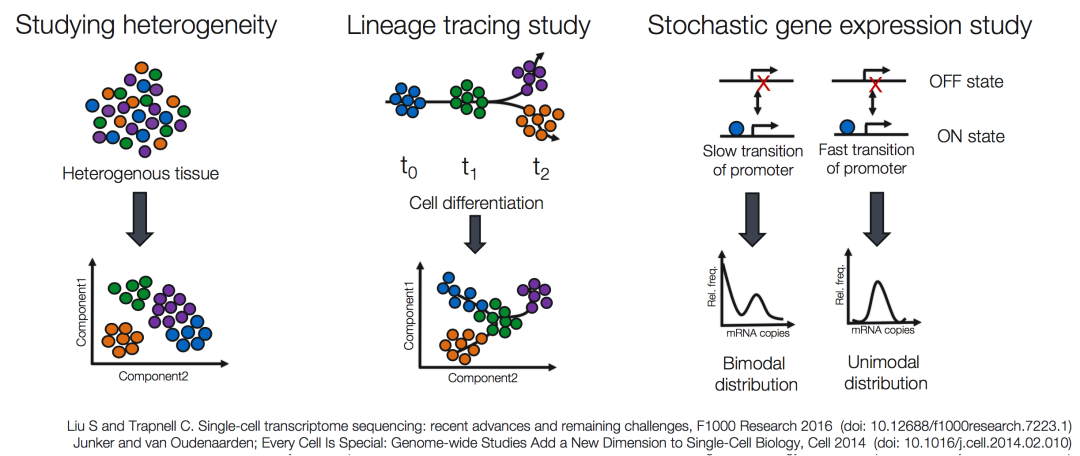
Challenges of scRNA-seq analysis
在scRNA-seq之前,我们通常使用bulk RNA-seq进行转录组分析。bulk RNA-seq是一种直接比较细胞的平均表达值的方法,在寻找疾病生物标志物,或者不关心样品中大量细胞异质性的情况下,这可能是最佳方法。
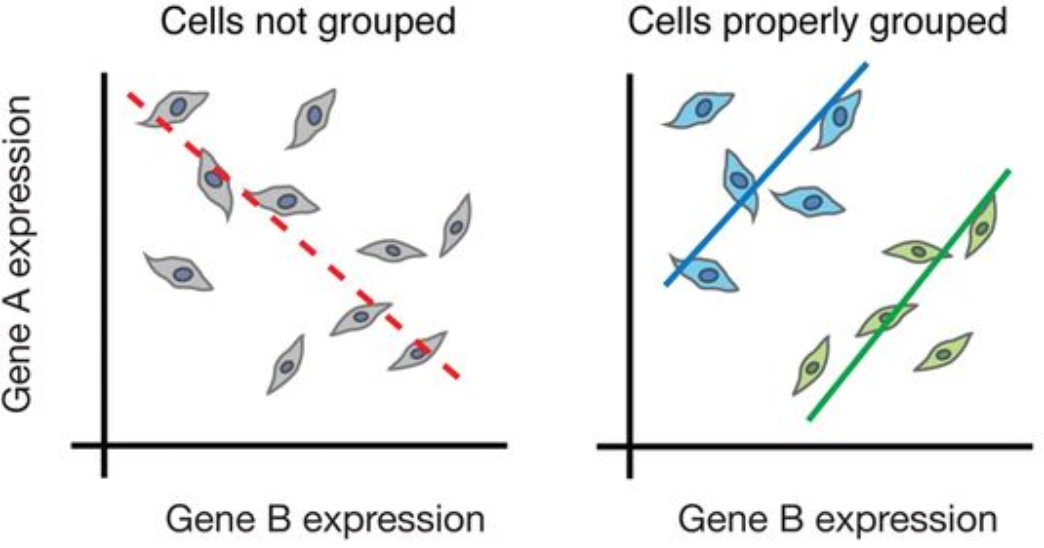
尽管bulk RNA-seq可以探索不同条件(例如治疗或疾病)之间基因表达的差异,但无法充分捕获细胞水平的差异。例如,在下面的图像中,如果进行bulk分析(左),我们将无法检测到基因A和基因B的表达之间的正确关联。但是,如果我们按细胞类型或细胞状态正确地对细胞进行分组,我们可以看到基因之间的正确相关性。
scRNA-seq也有一定的局限性,除了制样和建库价格高昂外,它在数据分析中也具有一定的复杂性,包括:
数据量大
细胞的测序深度低
细胞/样品之间的技术差异
跨细胞/样品的生物变异性
数据量大
scRNA-seq实验的数据来自捕获的成千上万甚至百万个细胞,对应的测序数据也就有百千万条reads,需要更多的内存和存储空间。
细胞的测序深度低
基于液滴的scRNA-seq方法的测序深度较浅,通常每个细胞只能检测到转录组的10-50% (生信宝典注:这主要是因为RNA捕获率低)。这导致细胞中许多基因的计数为零 (可能是不表达,也可能是未捕获到)。但是,在特定的细胞中,基因的零计数可能意味着该基因未表达或仅表示该转录本未被检测到。在整个细胞中,具有较高表达水平的基因测到0值的机率较低。由于此特征,在任何细胞中都不会检测到全部基因,并且细胞之间的基因表达差异很大。
Zero-inflated?:scRNA-seq data is often referred to as zero-inflated; however, recent analyses suggest that it does not contain more zeros than what would be expected given the sequencing depth (Valentine Svensson’s blog post:http://www.nxn.se/valent/2017/11/16/droplet-scrna-seq-is-not-zero-inflated).
跨细胞/样品的生物变异性
我们不感兴趣的某些生物差异可能导致细胞之间的基因表达比实际生物细胞的类型/状态更为相似/不同,并掩盖细胞类型。这些变异(除非实验研究的一部分)包括(以下为原文):
Transcriptional bursting: Gene transcription is not turned on all of the time for all genes. Time of harvest will determine whether gene is on or off in each cell.
Varying rates of RNA processing: Different RNAs are processed at different rates.
Continuous or discrete cell identities (e.g. the pro-inflammatory potential of each individual T cell): Continuous phenotypes are by definitition variable in gene expression, and separating the continuous from the discrete can sometimes be difficult.
Environmental stimuli: The local environment of the cell can influence the gene expression depending on spatial position, signaling molecules, etc.
Temporal changes: Fundamental fluxuating cellular processes, such as cell cycle, can affect the gene expression profiles of individual cells.
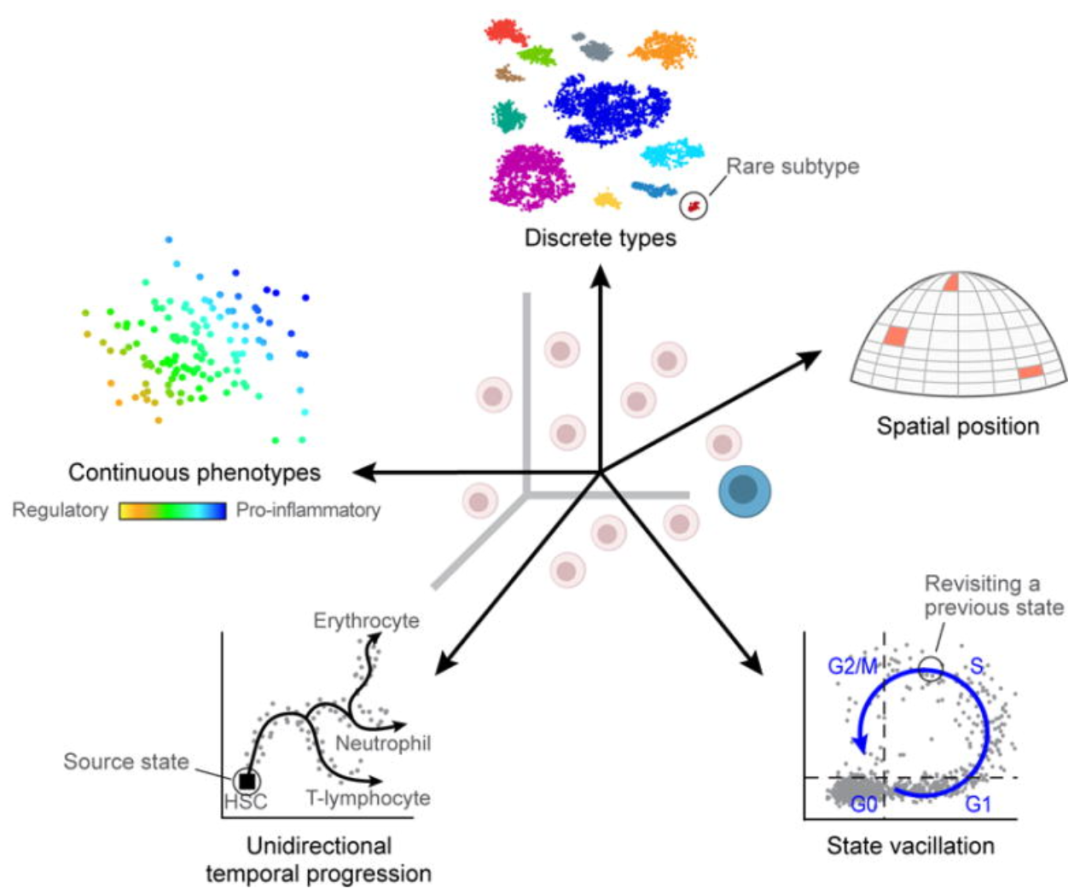
Image credit: Wagner, A, et al. Revealing the vectors of cellular identity with single-cell genomics, Nat Biotechnol. 2016 (doi:https://dx.doi.org/10.1038%2Fnbt.3711)
细胞/样品之间的技术差异
细胞特异性捕获效率:
不同细胞捕获的转录本数量不同,导致测序深度不同(例如,转录组的10-50%)。
文库质量:
降解的RNA、低存活力/濒死细胞、大量自由漂浮的RNA以及细胞定量不准确会导致质量指标降低。
扩增偏差:
在文库制备的扩增步骤中,并非所有转录本都扩增相同次数。
批次效应:
对于scRNA-Seq分析,批次效应是一个重要的问题,因为看到的显著差异表达可能只是因为批次效应引起的。
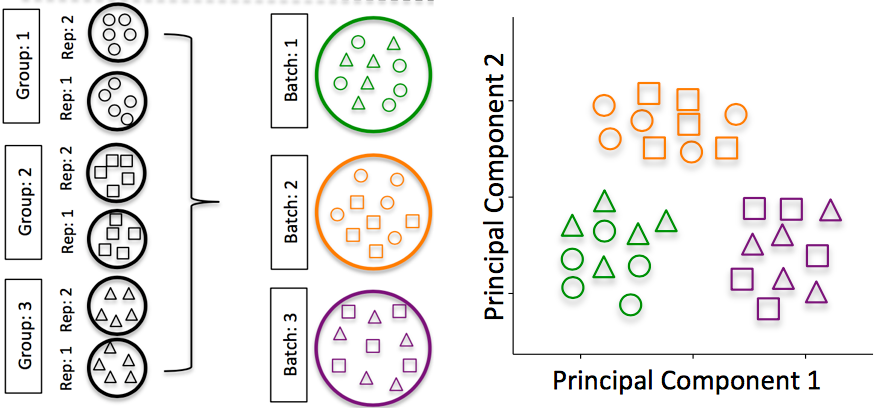
Image credit: Hicks SC, et al., bioRxiv (2015)(https://www.biorxiv.org/content/early/2015/08/25/025528)_
批次不良所产生的问题在这篇文章中有很好的介绍:https://f1000research.com/articles/4-121/v1,相关中文解读见高通量数据中批次效应的鉴定和处理 - 系列总结和更新。
如何知道你的实验中具有批次呢?
是否在同一天进行了所有RNA的分离?
是否在同一天进行了所有建库工作?
是否由同一个人对所有样品进行RNA分离/文库制备?
是否对所有样品使用相同的试剂?
是否在同一位置进行RNA分离/文库制备?
如果以上任一问题的答案为“否”,说明你的实验中具有批次。
有关批次的最佳做法:
尽可能避免以批次的方式设计实验。
如果无法避免:
Do NOT confound your experiment by batch:
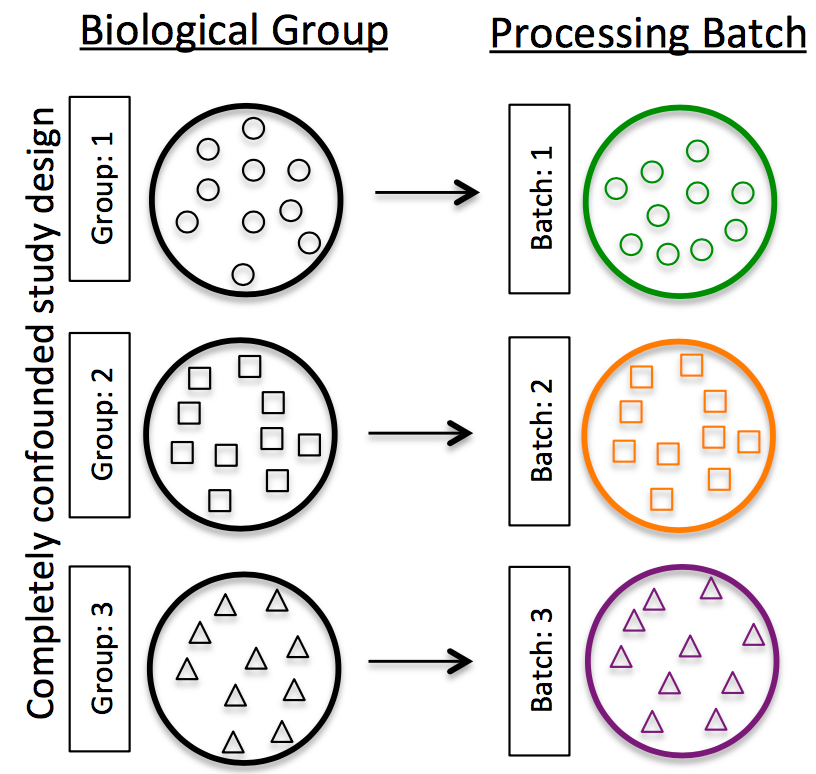
将不同样品组的重复样品分成多个批次:
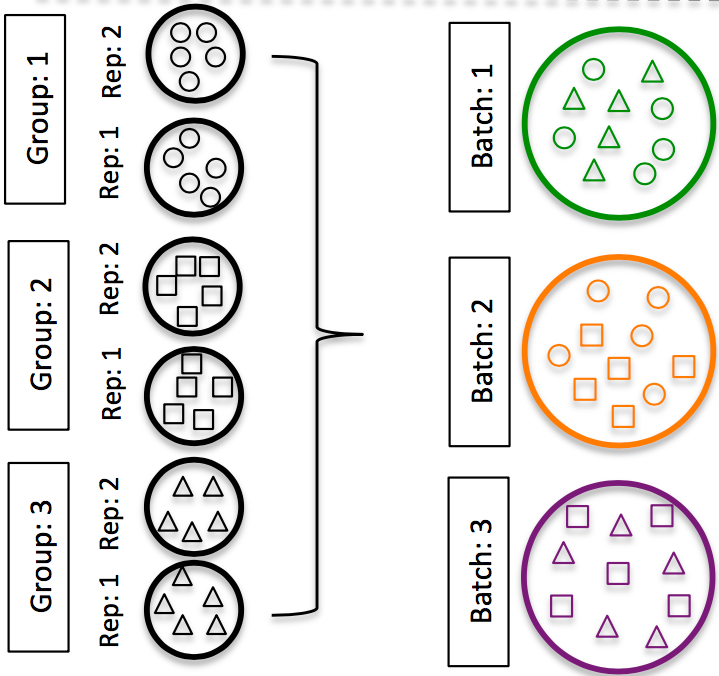
在实验设计文件中加上批次信息,这样可以在分析过程中退还批次引起的差异。
建议:
在实验开始之前与专家讨论实验设计。
同时从样品中分离RNA。
同时准备样品库或备用样品组,以避免批次混淆。
不要混淆性别、年龄或批次的样本组。
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集